Powszechnie wiadomo, że w głównym dogmacie RNA jest mediatorem transkrypcji między DNA a ekspresją białek.W porównaniu z wykrywaniem DNA, wykrywanie RNA może bardziej obiektywnie odzwierciedlać ekspresję genów w organizmach.Eksperymenty z udziałem RNA obejmują: qRT-PCR, RNA-Seq i wykrywanie genów fuzyjnych itp. Opierając się na charakterystyce samego RNA (pierścień cukrowy RNA ma o jedną wolną grupę hydroksylową więcej niż pierścień cukrowy DNA), w połączeniu z dużą liczbą RNaz w środowisku, RNA jest bardziej niestabilny i łatwiej ulega degradacji niż DNA.Śmieci na wejściu, śmieci na wyjściu, jeśli jakość RNA nie jest dobra, wyniki eksperymentów muszą być niezadowalające, co objawia się w szczególności niedokładnymi danymi lub słabą powtarzalnością.Dlatego też należy zwrócić większą uwagę na przetwarzanie RNA, a powiązanie kontroli jakości jest również ważniejsze dla zapewnienia precyzji i dokładności kolejnych danych eksperymentalnych.
Do kontroli jakości RNA ogólnie stosuje się następujące powszechnie stosowane metody:
- Spektrofotometria
- elektroforeza w żelu agarozowym
- Bioanalizator Agilent
- fluorescencyjny ilościowy PCR w czasie rzeczywistym
- Metoda barwnika fluorescencyjnego qubit
01 Spektrofotometria
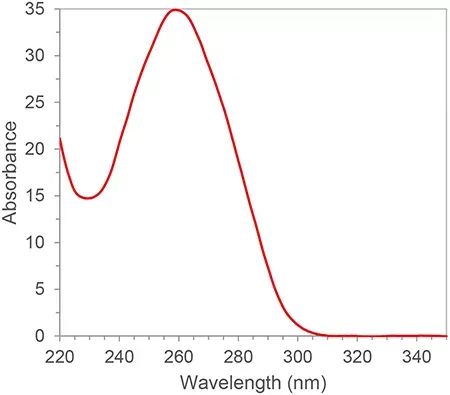
RNA ma sprzężone wiązania podwójne i ma pik absorpcji przy długości fali 260 nm.Zgodnie z prawem Lamberta-Beera stężenie RNA możemy obliczyć z piku absorpcji przy 260 nm.Ponadto możemy również obliczyć czystość RNA według stosunku pików absorpcji 260nm, 280nm i 230nm.280nm i 230nm to odpowiednio szczyty absorpcji białek i małych cząsteczek.Stosunek A260/A280 i A260/A230 kwalifikowanej czystości RNA powinien być większy niż 2. Jeśli jest mniejszy niż 2, oznacza to, że w próbce RNA występuje zanieczyszczenie białkiem lub drobnocząsteczką i należy ją ponownie oczyścić.Źródła zanieczyszczeń wpłyną na późniejsze eksperymenty, takie jak hamowanie wydajności amplifikacji reakcji PCR, co skutkuje niedokładnymi wynikami ilościowymi.Czystość RNA ma ogromny wpływ na późniejsze wyniki, dlatego spektrofotometria jest ogólnie niezbędnym ogniwem kontroli jakości na pierwszym etapie eksperymentów z kwasami nukleinowymi.
Rysunek 1. Typowe widmo absorpcji RNA/DNA
02 Elektroforeza w żelu agarozowym
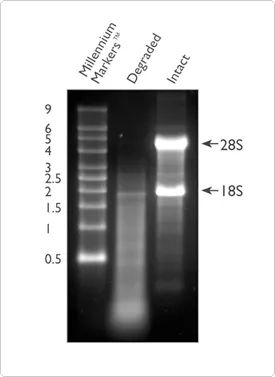
Oprócz czystości integralność RNA jest również jednym z ważnych wskaźników oceny jakości RNA.Degradacja RNA doprowadzi do dużej liczby krótkich fragmentów w próbce, więc zmniejszy się liczba fragmentów RNA, które można skutecznie wykryć i pokryć sekwencją referencyjną.Integralność RNA można sprawdzić za pomocą elektroforezy całkowitego RNA na 1% żelu agarozowym.Ta metoda umożliwia samodzielne skonfigurowanie żelu lub użycie prefabrykowanego systemu E-Gel™ do testowania integralności.Ponad 80% całkowitego RNA to rybosomalny RNA, z którego większość składa się z 28S i 18S rRNA (u ssaków).Dobrej jakości RNA pokaże dwa oczywiste jasne słupki, które są odpowiednio jasnymi słupkami 28S i 18S, przy 5 Kb i 2 Kb, a stosunek będzie bliski 2:1.Jeśli jest w stanie rozproszonym, oznacza to, że próbka RNA mogła ulec degradacji i zaleca się zastosowanie metody opisanej w dalszej części badania jakości RNA.
Rycina 2. Porównanie zdegradowanego (ścieżka 2) i nienaruszonego RNA (ścieżka 3) w elektroforezie w żelu agarozowym
03 Bioanalizator Agilent
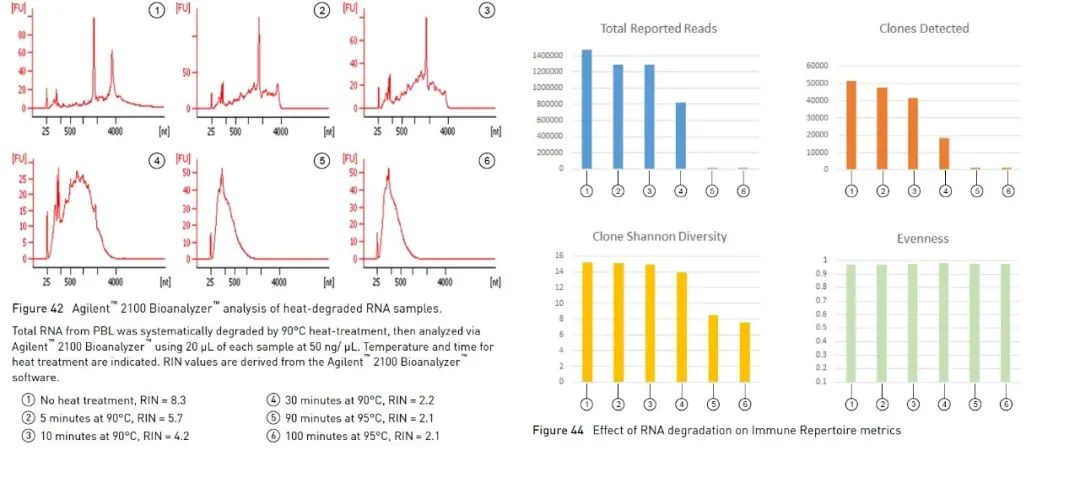
Oprócz opisanej powyżej metody elektroforezy w żelu agarozowym, która może pomóc nam w prosty i szybki sposób zidentyfikować integralność RNA, możemy również użyć bioanalizatora Agilent do określenia integralności RNA.Wykorzystuje kombinację mikroprzepływów, elektroforezy kapilarnej i fluorescencji do oceny stężenia i integralności RNA.Korzystając z wbudowanego algorytmu do analizy profilu próbki RNA, bioanalizator Agilent może obliczyć referencyjną wartość integralności RNA, RNA Integrity Number (zwaną dalej RIN) [1].Im większa wartość RIN, tym wyższa integralność RNA (1 oznacza skrajnie zdegradowany, 10 najbardziej kompletny).Niektóre eksperymenty z udziałem RNA sugerują użycie RIN jako parametru do oceny jakości.Biorąc za przykład wysokoprzepustowe eksperymenty sekwencjonowania (zwane dalej NGS), wytyczne Oncomine™ Human Immune Repertoire, które są wykorzystywane do wykrywania receptorów antygenów limfocytów B i T w serii paneli Oncomine firmy Thermo Fisher, sugerują, że próbki o wartościach RIN większych niż 4, można mierzyć bardziej efektywne odczyty i klony (Rysunek 3).Istnieją różne zalecane zakresy dla różnych paneli i często wyższy RIN może przynieść bardziej efektywne dane.
Rysunek 3, w doświadczeniach Oncomine™ Human Immune Repertoire, próbki z RIN większym niż 4 mogą wykrywać skuteczniejsze odczyty i klony komórek T.【2】
Jednak wartość RIN ma również pewne ograniczenia.Chociaż RIN ma wysoką korelację z jakością danych eksperymentalnych NGS, nie nadaje się do próbek FFPE.Próbki FFPE były przez długi czas poddawane obróbce chemicznej, a wyekstrahowany RNA na ogół ma stosunkowo niską wartość RIN.Nie oznacza to jednak, że efektywne dane eksperymentu muszą być niezadowalające.Aby dokładnie ocenić jakość próbek FFPE, musimy zastosować pomiary inne niż RIN.Oprócz RIN, bioanalizator Agilent może również obliczyć wartość DV200 jako parametr oceny jakości RNA.DV200 to parametr, który oblicza odsetek fragmentów większych niż 200 bp w próbce RNA.DV200 jest lepszym wskaźnikiem jakości próbki FFPE niż RIN.W przypadku RNA wyekstrahowanego metodą FFPE ma on bardzo wysoką korelację z liczbą genów, które można skutecznie wykryć oraz różnorodnością genów [3].Chociaż DV200 może nadrobić braki w wykrywaniu jakości FFPE, bioanalizator Agilent nadal nie może kompleksowo analizować problemów jakościowych w próbkach RNA, w tym czy w próbkach znajdują się inhibitory.Same inhibitory mogą wpływać na wydajność amplifikacji dalszych eksperymentów i zmniejszać ilość użytecznych danych.Aby wiedzieć, czy w próbce znajduje się inhibitor, możemy zastosować opisaną poniżej metodę ilościowej fluorescencyjnej reakcji PCR w czasie rzeczywistym.
04 fluorescencyjny ilościowy PCR w czasie rzeczywistym
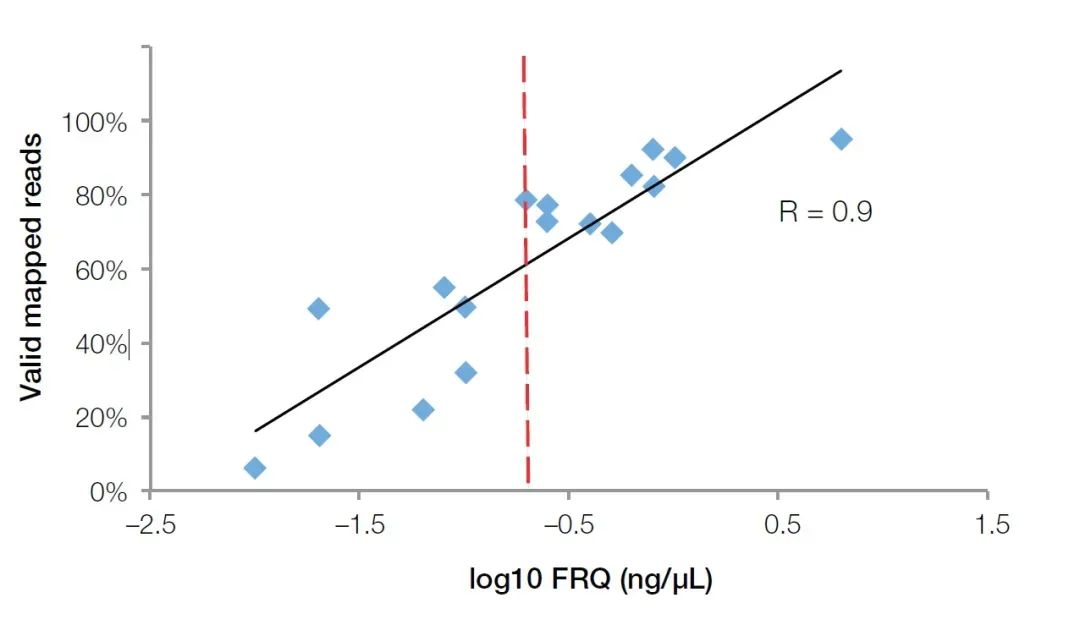
Ilościowa metoda fluorescencyjnej PCR w czasie rzeczywistym może nie tylko wykryć inhibitory w próbce, ale także dokładnie odzwierciedlać jakość RNA w próbce FFPE.W porównaniu z analizatorami biologicznymi Agilent, ilościowe instrumenty fluorescencji w czasie rzeczywistym są bardziej popularne w dużych laboratoriach biologicznych ze względu na ich szersze zastosowanie.Aby zbadać jakość próbek RNA, wystarczy zakupić lub przygotować sondy starterowe dla wewnętrznych genów referencyjnych, takich jak GUSB (nr kat. Hs00939627).Używając tego zestawu starterów, sond i wzorców (całkowity RNA o znanym stężeniu) do przeprowadzania bezwzględnych eksperymentów ilościowych, efektywne stężenie fragmentów RNA można obliczyć jako standard oceny jakości RNA (w skrócie Functional RNA Quantitation (FRQ)).W teście NGS odkryliśmy, że FRQ próbek RNA ma bardzo wysoką korelację z efektywną objętością danych.Dla wszystkich próbek powyżej 0,2 ng/uL FRQ co najmniej 70% odczytów może skutecznie pokryć sekwencję referencyjną (Rysunek 4).
Na rycinie 4 wartość FRQ wykryta metodą ilościową fluorescencji ma bardzo wysoką korelację (R2>0,9) z danymi efektywnymi uzyskanymi w eksperymencie NGS.Czerwona linia to wartość FRQ równa 0,2 ng/uL (log10 = -0,7).【4】
Oprócz możliwości zastosowania do próbek FFPE, ilościowa metoda PCR w czasie rzeczywistym może również skutecznie monitorować inhibitory w próbkach.Możemy dodać próbkę, która ma być wykryta, do układu reakcyjnego z wewnętrzną kontrolą pozytywną (IPC) i jej oznaczeniem, a następnie przeprowadzić ocenę ilościową fluorescencji w celu uzyskania wartości Ct.Jeśli wartość Ct pozostaje w tyle za wartością Ct w reakcji bez próbki, oznacza to, że inhibitor jest obecny w próbce i hamuje wydajność amplifikacji w reakcji.
05 Metoda barwnika fluorescencyjnego Qubit
Fluorometr Qubit to najczęściej używane małe urządzenie do wykrywania stężenia i czystości kwasów nukleinowych, które jest łatwe w obsłudze i znajduje się w prawie każdym laboratorium biologii molekularnej.Dokładnie oblicza stężenie kwasu nukleinowego, wykrywając i wiążąc kwas nukleinowy barwnik fluorescencyjny (odczynnik do wykrywania Qubit).Qubit ma wysoką czułość i specyficzność i może dokładnie określić ilościowo RNA aż do stężenia pg/µL.Oprócz dobrze znanej zdolności do dokładnego ilościowego określania stężenia kwasu nukleinowego, najnowszy nowy model firmy Thermo Fisher, Qubit 4.0, może również wykrywać integralność RNA.System wykrywania RNA Qubit 4.0 (RNA IQ Assay) wykrywa integralność RNA poprzez jednoczesne wykrywanie dwóch specyficznych barwników fluorescencyjnych.Te dwa barwniki fluorescencyjne mogą wiązać się odpowiednio z dużymi fragmentami i małymi fragmentami RNA.Te dwa barwniki fluorescencyjne wskazują proporcję dużych fragmentów RNA w próbce iz tego można obliczyć wartość IQ (integralność i jakość) reprezentującą jakość RNA.Wartość IQ ma zastosowanie zarówno do próbek FFPE, jak i innych niż FFPE i ma duży wpływ na późniejszą jakość sekwencjonowania.Biorąc za przykład eksperymenty NGS, w eksperymentach testowych RNA-Seq przeprowadzonych na platformie Ion torrent™ większość próbek z wartościami IQ większymi niż 4 miała co najmniej 50% skutecznych odczytów (Rysunek 5).W porównaniu z wyżej wymienionymi metodami wykrywania, Qubit IQ Assay jest nie tylko wygodniejszy w obsłudze i zajmuje mniej czasu (w ciągu pięciu minut), ale ma również doskonałą korelację między wartością mierzonego parametru IQ a jakością danych dalszych eksperymentów.
Ryc. 5, istnieje duża korelacja między wartością Qubit RNA IQ a zmapowanymi odczytami RNA-Seq.【5】
Wierzę, że dzięki powyższemu wstępowi każdy ma wystarczającą wiedzę na temat różnych metod kontroli jakości RNA.W praktyce możesz wybrać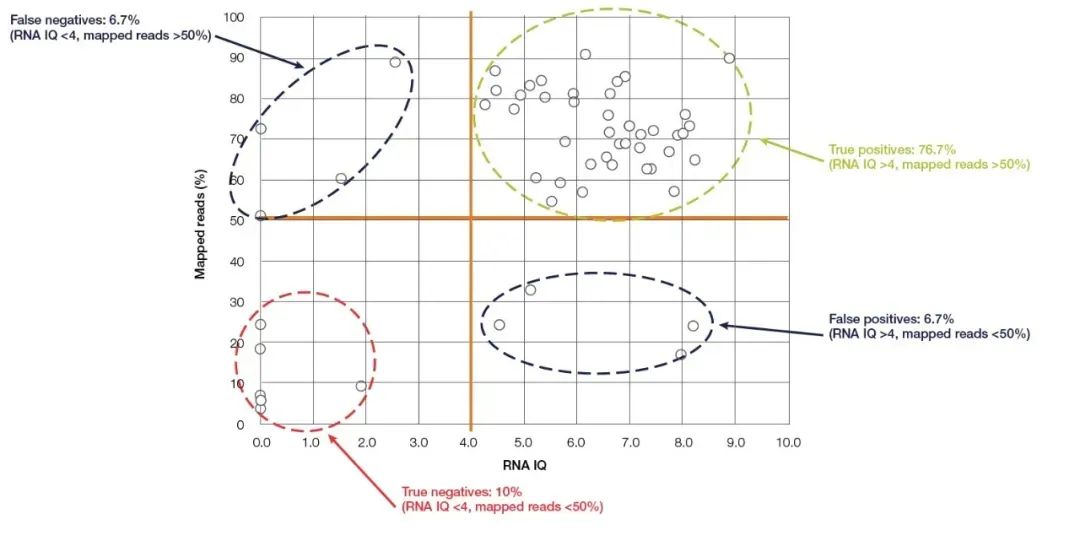 odpowiednią metodę w zależności od rodzaju próbki i istniejących instrumentów.Tylko dobrze kontrolując jakość RNA, możemy uniknąć niepowodzeń kolejnych eksperymentów spowodowanych złą jakością próbki, oszczędzając w ten sposób cenny czas, energię i koszty.
odpowiednią metodę w zależności od rodzaju próbki i istniejących instrumentów.Tylko dobrze kontrolując jakość RNA, możemy uniknąć niepowodzeń kolejnych eksperymentów spowodowanych złą jakością próbki, oszczędzając w ten sposób cenny czas, energię i koszty.
Produkty referencyjne:
Zestaw do izolacji całkowitego RNA zwierząt
Zestaw do izolacji całkowitego RNA komórek
Bibliografia
【1】Schroeder, A., Mueller, O., Stocker, S. et al.RIN: numer integralności RNA służący do przypisywania wartości integralności pomiarom RNA.BMC Molecular Biol 7, 3 (2006).https:// doi.org/10.1186/1471-21 99-7-3
【2】 Podręcznik użytkownika Oncomine Human Immune Repertoire (Pub. No. MAN0017438 Rev. C.0).
【3】Leah C Wehmas, Charles E Wood, Brian N Chorley, Carole L Yauk, Gail M Nelson, Susan D Hester, Enhanced Quality Metrics for Assessing RNA Derived From Archives Formalin-Fixed Paraffin-Embedded Tissue Samples, Toxicological Sciences, tom 170, wydanie 2, sierpień 2019 r., strony 357–373,https://doi.org/10.1093/toxsci/
Czas postu: 12 czerwca 2023 r