Eksperyment RT-qPCR obejmuje ekstrakcję RNA i ocenę jakości, odwrotną transkrypcję i qPCR trzy etapy, każdy krok ma wiele środków ostrożności, które szczegółowo przedstawimy poniżej.
Ⅰ.Ocena jakości RNA
W eksperymencie RT-qPCR po zakończeniu ekstrakcji RNA należy ocenić jakość RNA, a eksperyment kontrolny można przeprowadzić dopiero po jego zakwalifikowaniu.Metody oceny obejmują spektrofotometr, elektroforezę w żelu Agilent, analizę Agilent 2100, wśród których najczęściej stosowany jest spektrofotometr oraz wykrywanie metodą elektroforezy w żelu agarozowym.Należy zauważyć, że te dwie metody muszą być stosowane razem, aby zakończyć wykrywanie i analizę stężenia, czystości i integralności RNA, tak aby zapewnić jakość RNA.
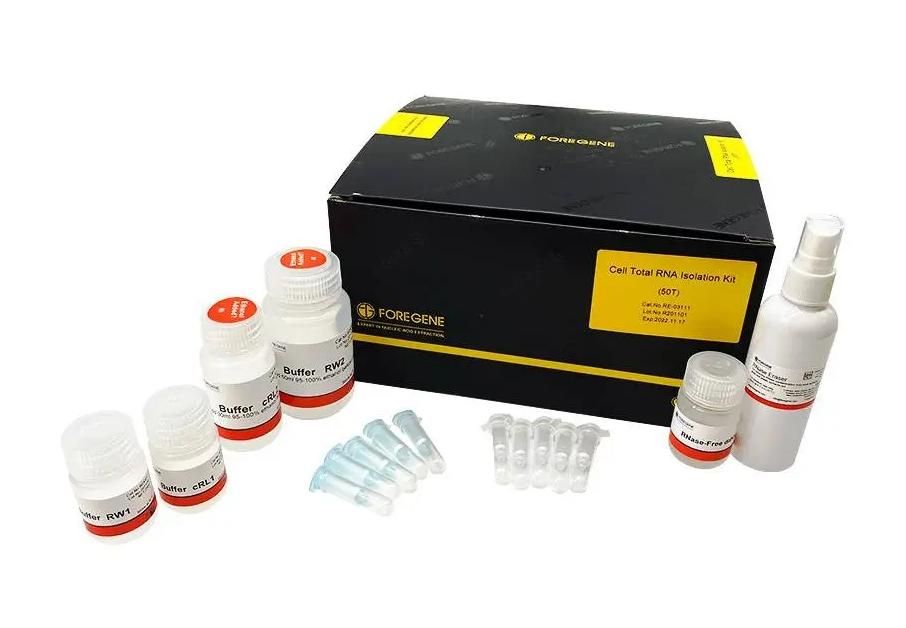
Powiązany zestaw do izolacji RNA:
Zestaw do izolacji całkowitego RNA komórek
Wysoce oczyszczony i wysokiej jakości całkowity RNA można uzyskać z różnych hodowanych komórek w ciągu 11 minut.
Zestaw do izolacji całkowitego RNA zwierząt
Szybko i wydajnie wyodrębnij całkowity RNA o wysokiej czystości i wysokiej jakości z różnych tkanek zwierzęcych.
Spektrofotometr:
Spektrofotometr służy głównie do określania stężenia i czystości RNA, ale nie może wykryć integralności RNA i pozostałości genomowych.Wśród nich A260/280 i A260/230 są ważnymi parametrami do wykrywania czystości RNA, a czystość RNA można wykryć na podstawie fluktuacji ich wartości:
1. 1,9 < A260/280 < 2,1, co wskazuje, że czystość RNA jest dobra;A260/280 <1,9, co wskazuje, że w RNA mogą znajdować się reszty białkowe;A260/280>2,1, co wskazuje na możliwą częściową degradację RNA, co można dalej potwierdzić za pomocą elektroforezy w żelu agarozowym.
2. 2,0 < A260/230 < 2,2, co wskazuje, że czystość RNA jest dobra;A260/230< 2,0, co wskazuje, że w RNA mogą znajdować się pozostałości odczynników organicznych, takich jak fenole, etanol czy cukry.
Elektroforeza w żelu agarozowym:
Test elektroforezy w żelu agarozowym może analizować integralność RNA, genom i reszty białkowe, ale nie może dokładnie określić ilościowo stężenia RNA ani wykryć pozostałości odczynników organicznych.Weźmy na przykład eukariotyczne szablony RNA:
1. RNA poddano elektroforezie w żelu agarozowym.Jeśli na mapie żelowej były tylko trzy pojedyncze prążki 28sRNA, 18sRNA i 5,8sRNA, oznacza to, że wyekstrahowany RNA jest nienaruszony.Jeśli występuje zjawisko przeciągania, oznacza to częściową degradację RNA.
2. Jeśli między dziurą po kleju a prążkiem 28sRNA znajduje się pojedynczy jasny prążek, może to oznaczać pozostałości genomowego DNA.
3. Jeśli w otworze po kleju pojawiają się prążki, oznacza to, że mogą znajdować się w nich pozostałości białka i innych substancji wielkocząsteczkowych.
Ⅱ. Transkrypcja odwrotna
Po zakończeniu ekstrakcji RNA należy go odwrócić do cDNA do kolejnych eksperymentów, więc etap odwrócenia jest niezbędny.Odwrotna transkrypcja zostanie wprowadzona z selekcji odwrotnej transkryptazy i startera:
Selekcja odwrotnej transkryptazy:
Typowe odwrotne transkryptazy obejmują RTazę AMV i RTazę MMLV.RNaza H AMV RTase ma silną aktywność, krótką długość syntezy, małą ilość syntezy i dobrą stabilność termiczną (42 ~ 55 ℃).Aktywność RNazy H MMLV RTase jest słaba, długość syntezy jest długa, ilość syntezy jest wysoka, a stabilność termiczna jest słaba (37 ~ 42 ℃).
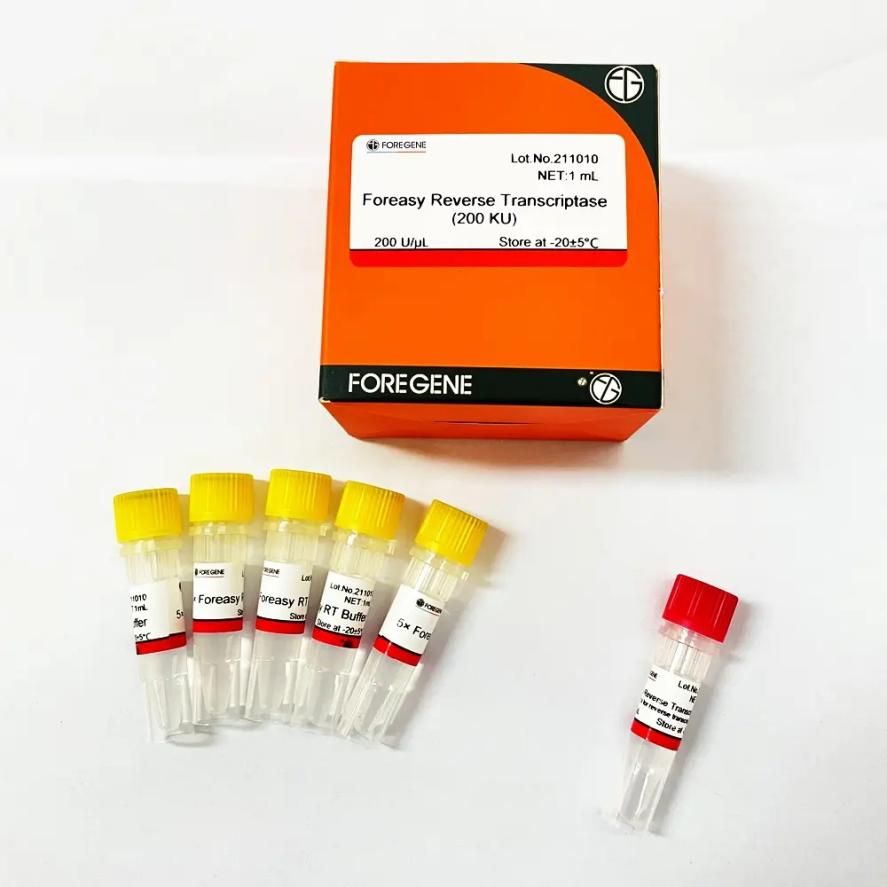
Ponieważ enzym RNaza H ma funkcję degradacji matrycy RNA, MMLV o słabej aktywności RNazy H powinien być preferencyjnie selekcjonowany podczas odwrotnej transkrypcji, a po późniejszej inżynierii genetycznej stabilność termiczna MMLV osiągnęła skok jakościowy.Biorąc Foregene'aForeasy Reverse Transcriptase (M-MLV dla odwrotnej transkrypcji) na przykład jest to nowa odwrotna transkryptaza eksprymowana w bakteriach E. coli modyfikowanych metodami inżynierii genetycznej przy użyciu technologii rekombinacji genetycznej.Jest to rekombinowana polimeraza DNA, która syntetyzuje komplementarną nić DNA z jednoniciowego RNA, DNA lub hybrydy RNA:DNA.Nie ma aktywności RNazy H, ma dużą stabilność, silne powinowactwo do RNA i wysoką czułość wykrywania.
Foreasy Reverse Transcriptase (M-MLV dla odwrotnej transkrypcji)
Wybór podkładu:
Ogólnie startery RT dzielą się na trzy kategorie: oligo dT, losowe startery i startery specyficzne dla genu.Wybierz odpowiednie startery do użycia zgodnie z różnymi wymaganiami eksperymentalnymi.
1. Jeśli matryca jest pochodzenia eukariotycznego, a późny cDNA jest używany do rutynowej amplifikacji PCR, zalecany jest Oligo (dT);Jeśli kolejny eksperyment jest używany tylko do qPCR, zaleca się zmieszanie Oligo (dT) z losowymi starterami w celu poprawy wydajności odwrotnej transkrypcji.
2. Jeśli matryca pochodzi od prokariotów, do odwrotnej transkrypcji należy wybrać losowe startery lub startery specyficzne dla genu.
Ⅲ.qPCR
Kwantyfikacja fluorescencji opiera się głównie na doborze metod ilościowych, zasadach projektowania starterów, wyborze ROX, konfiguracji systemu reakcji i ustawieniu warunków reakcji itp.
Wybór metod ilościowych:
Metody ilościowe dzielą się na względne metody ilościowe i bezwzględne metody ilościowe.Względne oznaczenie ilościowe można wykorzystać do wykrycia wpływu pewnych metod leczenia na ekspresję genów, wykrycia różnic w ekspresji genów w różnych momentach i porównania różnic w ekspresji genów w różnych tkankach.Bezwzględne oznaczenie ilościowe może wykryć ilość kwasu nukleinowego w wirusie i tak dalej.Wykonując eksperymenty, musimy wybrać odpowiednie metody ilościowe zgodnie z naszymi własnymi eksperymentami.
Zasady projektowania podkładu:
Projekt startera do qPCR jest bezpośrednio związany z wydajnością amplifikacji i specyficznością produktu.Dlatego też prawidłowe zaprojektowanie dobrych starterów jest pierwszym krokiem udanego qPCR.Podczas projektowania podkładu należy zwrócić uwagę na następujące zasady, spełniając zasadę konwencjonalnego projektowania podkładu:
1. Długość docelowego fragmentu jest kontrolowana w zakresie od 100 do 300 bp;
2. Projekt cross-exon, aby uniknąć wpływu genomowego DNA;
3. Zaprojektowane startery należy przetestować pod kątem wydajności amplifikacji i dopiero gdy skuteczność amplifikacji osiągnie standard (90-110%), można je wykorzystać do eksperymentów ilościowych;
4. Stężenie startera jest zwykle optymalizowane w zakresie od 0,1 uM do 1,0 uM.
WybórROX:
W procesie reakcji ilościowej ROX może równomiernie regulować różnicę dróg optycznych, błąd pipetowania lub różnicę objętości spowodowaną parowaniem i kondensacją, poprawiając powtarzalność wyników.Należy jednak zauważyć, że wybór ROX jest związany z instrumentem.Jeśli aparat qPCR posiada funkcję automatycznego korygowania różnicy między otworami, nie ma potrzeby dodawania ROX;w przeciwnym razie musi dodać korektę ROX.Mali partnerzy w zakupie odczynników muszą być zgodni z używanym instrumentem, aby wybrać właściwy ROX, aby uniknąć późniejszych błędów.
Przygotowanie układu reakcyjnego:
Korzystne są objętości reakcji 20 ul i 50 ul.Przy formułowaniu systemu należy zwrócić uwagę na następujące kwestie:
1. Układ reakcyjny należy przygotować poprzez wentylację w ultra czystym stole warsztatowym, nowy ddH2O jest używane w każdym eksperymencie;
2. Każdy eksperyment musi przygotować NTC, aby sprawdzić, czy w systemie nie ma zanieczyszczeń, a każda para starterów musi wykonać NTC podczas przygotowywania systemu;
3. Aby wykryć, czy w matrycy RNA znajduje się reszta gDNA, dla każdej próbki można przygotować NRT w celu wykrycia;
4. Przygotowując system zaleca się wykonanie co najmniej 3 powtórzeń technicznych dla jednej próbki;
5. Gdy matrycą jest cDNA, zaleca się 5-10-krotne rozcieńczenie, aby zredukować hamujący wpływ systemu odwrotnej transkrypcji na eksperyment qPCR.Lepiej jest badać ilość szablonów według gradientu, tak aby wartość CT mieściła się w przedziale 20-30;
6. Określ wymaganą liczbę reakcji, zwiększ ją o 5-10% na podstawie liczby reakcji i oblicz liczbę konfiguracji objętości;
7, system jest przygotowywany na zasadzie wstępnej mieszanki, mieszając po odwirowaniu i upewniając się, że nie ma pęcherzyków;
8, o ile to możliwe, aby wybrać wspierające materiały eksploatacyjne.
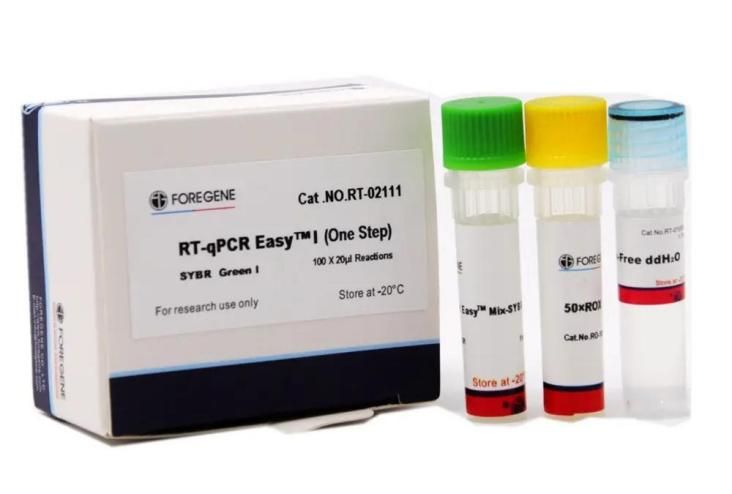
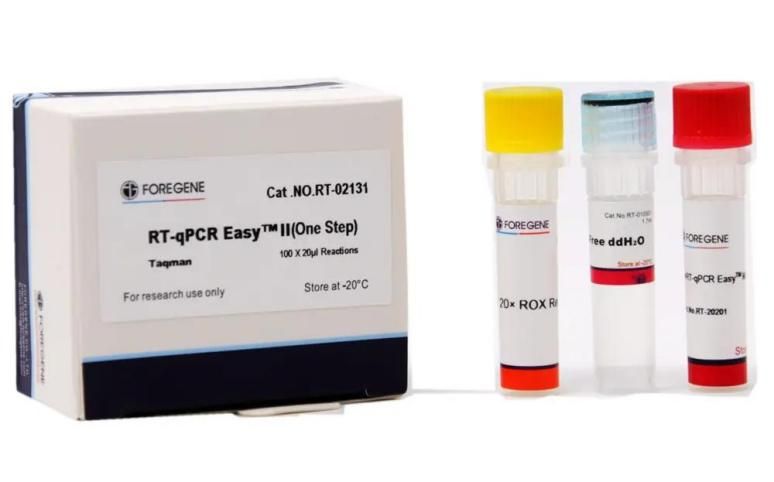
Powiązany zestaw RT-qPCR
Zestaw wykorzystuje unikalny odczynnik Foregene do odwrotnej transkrypcji i polimerazę Foregene HotStar Taq DNA w połączeniu z unikalnym systemem reakcji, aby skutecznie poprawić wydajność amplifikacji i specyficzność reakcji.
Czas postu: 23-04-2023