W eksperymentach qPCR projektowanie starterów jest również bardzo ważnym ogniwem.To, czy startery są odpowiednie, czy nie, jest ściśle związane z tym, czy wydajność amplifikacji osiąga standard, czy amplifikowane produkty są specyficzne i czy dostępne są wyniki eksperymentalne.
Jak więc poprawić specyficzność starterów qPCR?Wysoka wydajność wzmocnienia?
Dzisiaj zabierzemy Cię do wspólnego projektowania starterów qPCR i sprawimy, że projektowanie starterów qPCR stanie się skuteczną umiejętnością wiedzy w eksperymentach.
Projektując startery qPCR, zwykle należy zwrócić uwagę na następujące punkty: startery powinny być zaprojektowane w poprzek intronów w jak największym stopniu, długość produktu powinna wynosić 100-300 pz, wartość Tm powinna być jak najbliższa 60°C, a startery upstream i downstream powinny być jak najbliżej, a koniec startera powinien być G lub C itp. poczekaj.
1. Projektowanie starterów obejmujących introny
Podczas projektowania starterów qPCR wybór starterów zaprojektowanych w poprzek intronów może zapobiec amplifikacji matrycy gDNA, a wszystkie produkty pochodzą z amplifikacji cDNA, eliminując w ten sposób wpływ zanieczyszczenia gDNA.
2. Długość podkładu
Długość startera wynosi na ogół między 18-30 nt, a długość produktu amplifikacji powinna być w miarę możliwości kontrolowana między 100-300 bp.
Jeśli starter jest zbyt krótki, doprowadzi to do niespecyficznej amplifikacji, a jeśli jest zbyt długi, łatwo utworzy strukturę drugorzędową (taką jak struktura spinki do włosów).Jeśli produkt amplifikacji jest zbyt długi, nie nadaje się do reakcji polimerazy, co wpłynie na wydajność amplifikacji PCR.
3. Zawartość GC i wartość Tm
Zawartość GC starterów powinna być kontrolowana w zakresie od 40% do 60%.Jeśli jest zbyt wysoki lub zbyt niski, nie sprzyja zainicjowaniu reakcji.Zawartość GC starterów do przodu i do tyłu powinna być zbliżona do siebie, aby uzyskać tę samą wartość Tm i temperaturę przyłączania.
Wartość Tm powinna mieścić się w zakresie 55-65°C tak dalece, jak to możliwe, ogólnie około 60°C, a wartość Tm w górnym i dolnym biegu powinna być jak najbardziej zbliżona, korzystnie nie większa niż 4°C.
4. Unikaj wybierania A na końcu 3' startera
Gdy koniec 3' startera jest niedopasowany, występują duże różnice w wydajności syntezy różnych zasad.Gdy ostatnią zasadą jest A, może ona również inicjować syntezę łańcucha nawet w przypadku niedopasowania, a gdy ostatnią zasadą jest T Gdy , wydajność indukcji niedopasowania jest znacznie zmniejszona.Dlatego staraj się unikać wybierania A na końcu 3′ startera i lepiej wybrać T.
Jeśli jest to starter sondy, koniec 5' sondy nie może być G, ponieważ nawet gdy pojedyncza zasada G jest połączona z fluorescencyjną grupą reporterową FAM, G może również stłumić sygnał fluorescencyjny emitowany przez grupę FAM, powodując wyniki fałszywie ujemne.Pojawić się.
5. Dystrybucja bazowa
Rozmieszczenie czterech zasad w starterze jest korzystnie losowe, unikając więcej niż 3 kolejnych G lub C na końcu 3' i więcej niż 3 kolejnych zasadG lub C są łatwe do wygenerowania parowania w regionie sekwencji bogatym w GC.
6. Obszar projektu startera powinien unikać złożonych struktur drugorzędowych.
Struktura drugorzędowa utworzona przez pojedynczą nić produktu amplifikacji wpłynie na płynny przebieg reakcji PCR.Przewidując z góry, czy w sekwencji docelowej występuje struktura drugorzędowa, staraj się unikać tego regionu w projektowaniu starterów.
7. Same startery i między starterami powinny starać się unikać kolejnych komplementarnych zasad.
Nie może być komplementarności kolejnych 4 zasad między samym starterem a starterem.Sam podkład nie powinien mieć komplementarnej sekwencji, w przeciwnym razie zwinie się, tworząc strukturę szpilki do włosów, co wpłynie na wyżarzającą kombinację podkładu i szablonu.
Sekwencje komplementarne nie mogą istnieć między starterami w górę iw dół.Komplementarność między starterami spowoduje powstanie dimerów starterów, co zmniejszy wydajność PCR, a nawet wpłynie na dokładność ilościową.Jeśli struktury primer-dimer i spinki do włosów są nieuniknione, wartość △G nie powinna być zbyt wysoka (powinna być mniejsza niż 4,5 kcal/mol).
8. Startery wzmacniają docelowy specyficzny produkt.
Ostatecznym celem wykrywania qPCR jest zrozumienie obfitości docelowego genu.Jeśli wystąpi niespecyficzna amplifikacja, oznaczanie ilościowe będzie niedokładne.Dlatego po zaprojektowaniu starterów należy je przetestować metodą BLAST, a specyficzność produktów porównać w bazie danych sekwencji.
Następnie bierzemy ludzki gen GAS6 (specyficzny dla zatrzymania wzrostu 6) jako przykład do zaprojektowania starterów qPCR.
01 gen zapytania
Homo GAS6za pośrednictwem NCBI.Tutaj powinniśmy zwrócić uwagę na porównanie nazwy genu i gatunku, aby upewnić się, że są one spójne.
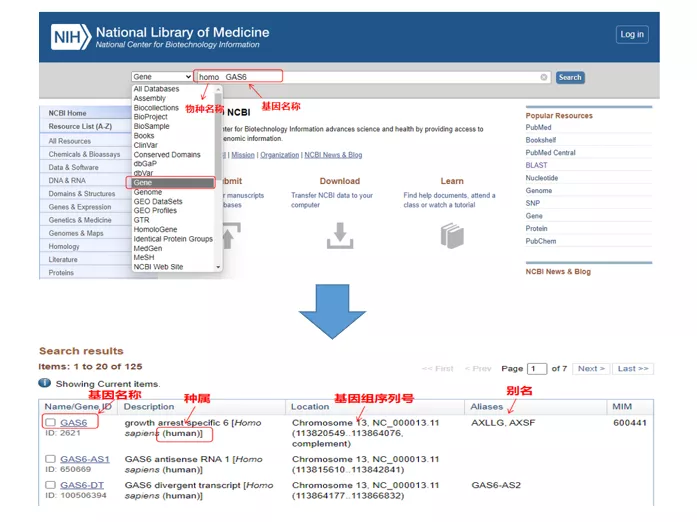 02 Znajdź sekwencję genu
02 Znajdź sekwencję genu
(1) Jeśli sekwencją docelową jest genomowy DNA, wybierz pierwszą, która jest sekwencją genomowego DNA genu.
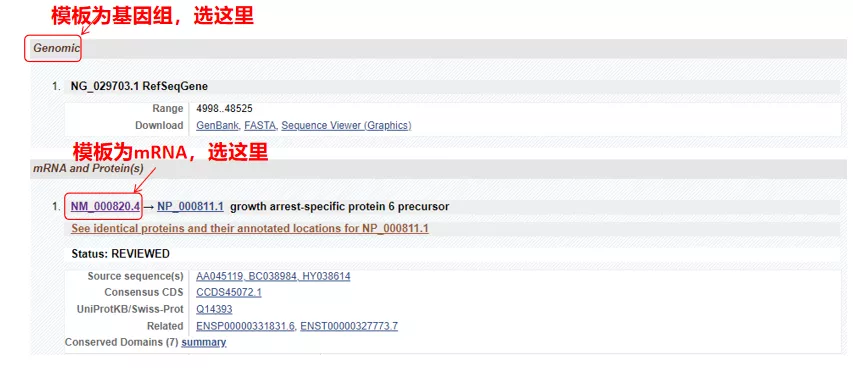 (2) Jeśli docelową sekwencją jest mRNA, wybierz drugą.Po wejściu kliknij „CDS” w poniższej tabeli.Brązowa sekwencja tła to sekwencja kodująca genu.
(2) Jeśli docelową sekwencją jest mRNA, wybierz drugą.Po wejściu kliknij „CDS” w poniższej tabeli.Brązowa sekwencja tła to sekwencja kodująca genu.
 03 Podkłady projektowe
03 Podkłady projektowe
Wejdź do interfejsu Primer-BLAST
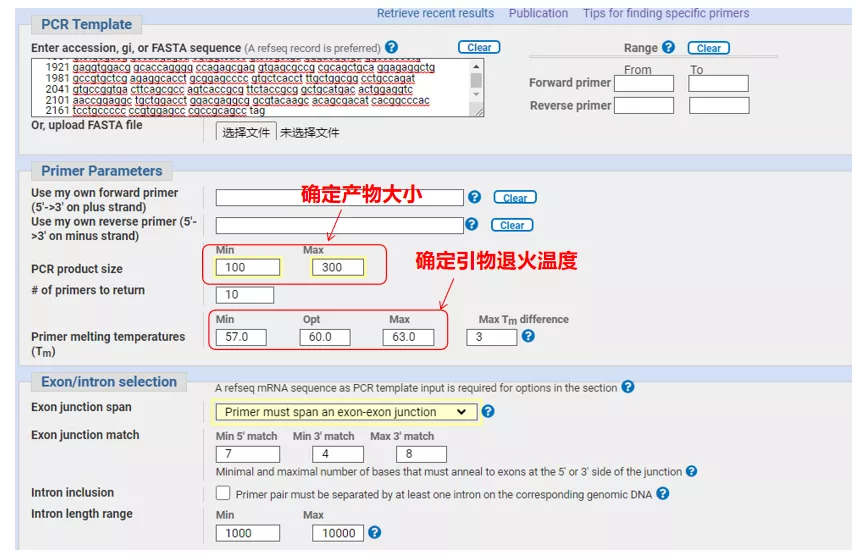
 Wprowadź numer sekwencji genu lub sekwencję w formacie Fasta w lewym górnym rogu i wypełnij odpowiednie parametry.
Wprowadź numer sekwencji genu lub sekwencję w formacie Fasta w lewym górnym rogu i wypełnij odpowiednie parametry.
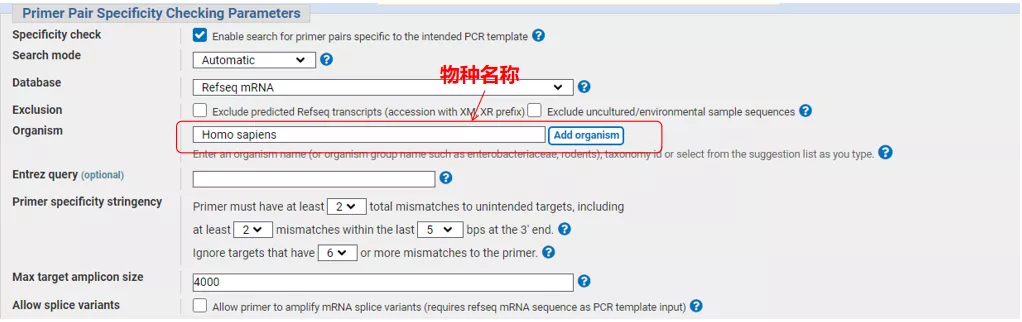
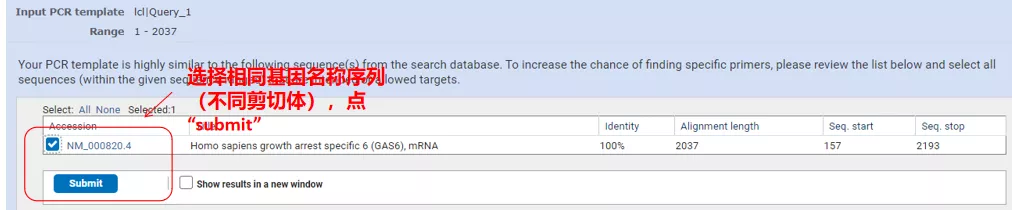
Kliknij „Get primers”, a NCBI wyskoczy z informacją, że taki wybór parametrów zostanie wzmocniony dla innych wariantów splicingu.Możemy sprawdzić różne warianty splicingu i przesłać je, aby uzyskać odpowiednią parę starterów (jak pokazano na poniższym rysunku).Uruchomienie tego procesu może zająć kilkadziesiąt sekund.
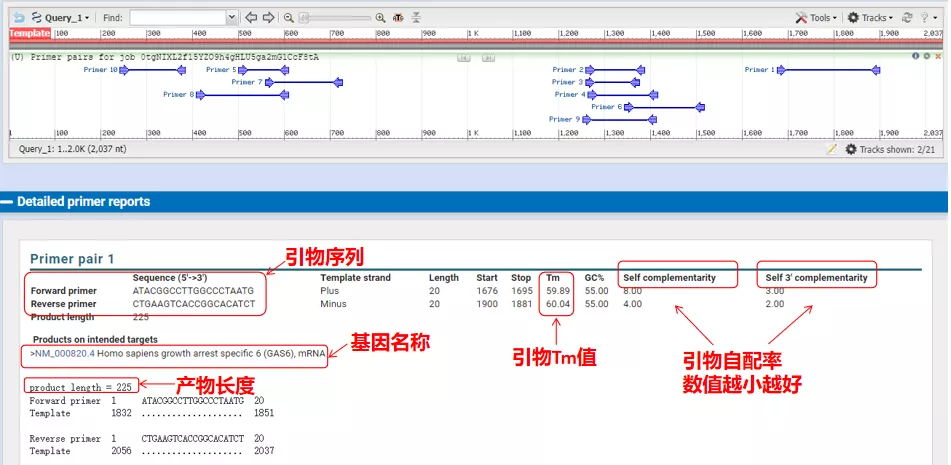 Temperatury wyżarzania tych par starterów wynoszą około 60°C.Zgodnie z celem eksperymentu wybierz startery o średniej długości, dobrej specyficzności i mniejszej samouzupełnianiu się starterów do eksperymentu, a wskaźnik sukcesu jest dość wysoki!
Temperatury wyżarzania tych par starterów wynoszą około 60°C.Zgodnie z celem eksperymentu wybierz startery o średniej długości, dobrej specyficzności i mniejszej samouzupełnianiu się starterów do eksperymentu, a wskaźnik sukcesu jest dość wysoki!
04Weryfikacja specyficzności startera
W rzeczywistości, oprócz projektowania podkładów, Primer-Blast może również oceniać podkłady, które sami zaprojektowaliśmy.Wróć do strony projektu podkładu, wprowadź zaprojektowane przez nas startery górny i dolny, a inne parametry nie zostaną dostosowane.Po przesłaniu można zobaczyć, czy para starterów występuje również na innych genach.Jeśli wszystkie z nich są wyświetlane na genie, który chcemy amplifikować, wskazuje to, że specyficzność tej pary starterów jest świetna!(Na przykład jest to jedyny wynik zapytania o elementarz!)
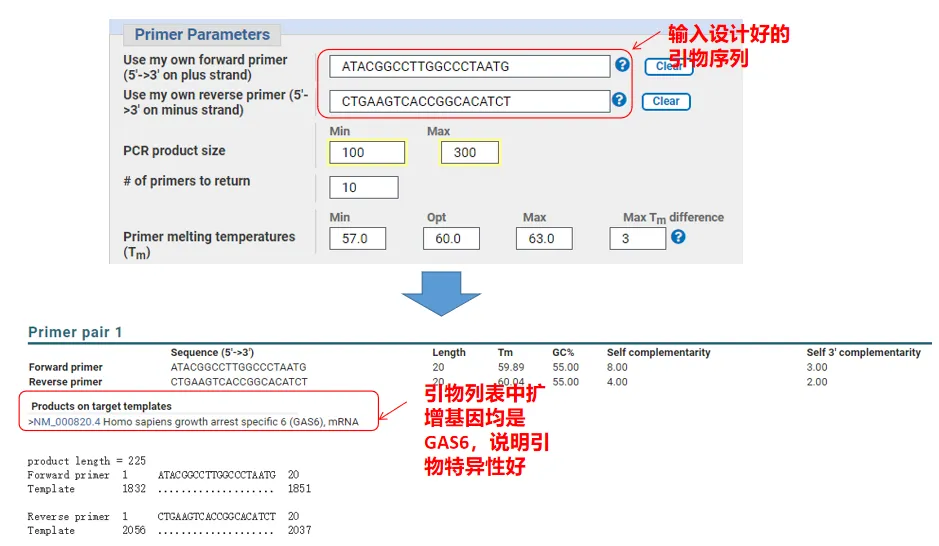
05 Ocena jakości podkładu
Jaki rodzaj startera jest „idealny”, który łączy w sobie „wydajność amplifikacji zgodną ze standardem”, „charakterystykę wzmocnionego produktu” i „wiarygodne wyniki eksperymentalne”?
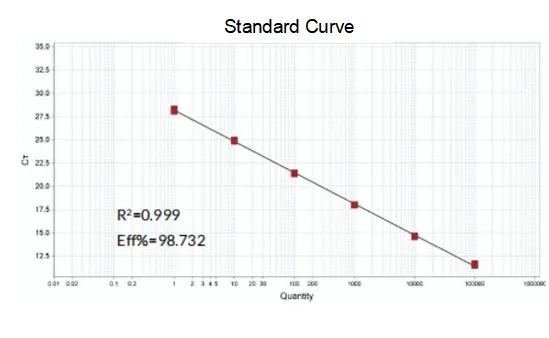 Wydajność amplifikacji
Wydajność amplifikacji
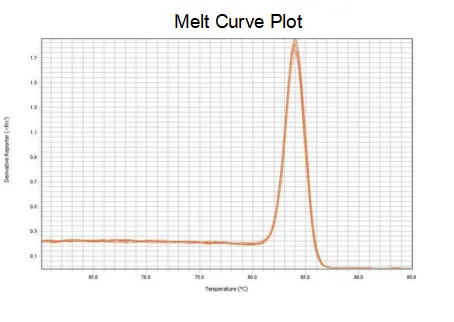 krzywa topnienia
krzywa topnienia
Wydajność amplifikacji starterów sięga 90%-110%, co oznacza, że wydajność amplifikacji jest dobra, a krzywa topnienia ma pojedynczy pik i zwykle Tm>80°C, co oznacza, że specyficzność amplifikacji jest dobra.
Produkty powiązane:
PCR w czasie rzeczywistym Easy–SYBR GREEN I
PCR w czasie rzeczywistym Easy-Taqman
Czas postu: 10 lutego 2023 r








