1. Wiedza podstawowa (jeśli chcesz zobaczyć część eksperymentalną, przejdź bezpośrednio do części drugiej)
Jako reakcja pochodna konwencjonalnego PCR, PCR w czasie rzeczywistym monitoruje głównie zmianę ilości produktu amplifikacji w każdym cyklu reakcji amplifikacji PCR w czasie rzeczywistym poprzez zmianę sygnału fluorescencji i analizuje ilościowo matrycę wyjściową poprzez związek między wartością ct a krzywą standardową.
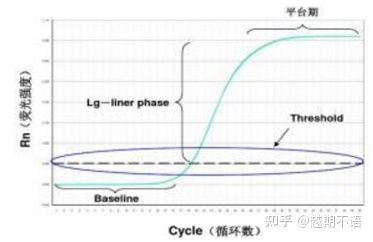
Specyficzne dane RT-PCR sąlinia bazowa, próg fluorescencjiIWartość Ct.
| linia bazowa: | Wartość fluorescencji 3-15 cyklu jest linią podstawową (bazową), która jest spowodowana sporadycznym błędem pomiaru. |
| Próg (próg): | Odnosi się do granicy wykrywalności fluorescencji ustawionej w odpowiedniej pozycji w obszarze wzrostu wykładniczego krzywej amplifikacji, zwykle 10-krotności odchylenia standardowego linii bazowej. |
| wartość przekładnika prądowego: | Jest to liczba cykli PCR, po których wartość fluorescencji w każdej probówce reakcyjnej osiągnie wartość progową. Wartość Ct jest odwrotnie proporcjonalna do ilości początkowej matrycy. |
Typowe metody znakowania dla RT-PCR:
| metoda | korzyść | niedociągnięcie | szereg zastosowań |
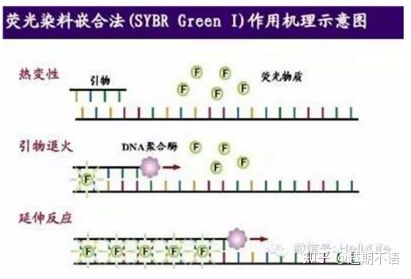
| SYBR ZielonyⅠ | Szerokie zastosowanie, czułe, tanie i wygodne | Wymagania dotyczące podkładu są wysokie, podatne na niespecyficzne prążki | Nadaje się do analizy ilościowej różnych genów docelowych, badań nad ekspresją genów oraz badań nad transgenicznymi rekombinowanymi zwierzętami i roślinami. |
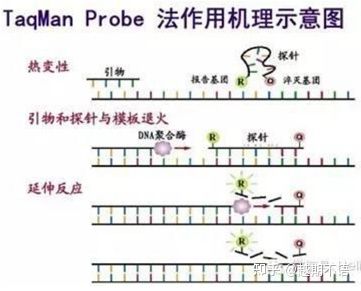
| TaqMan | Dobra specyficzność i wysoka powtarzalność | Cena jest wysoka i nadaje się tylko do określonych celów. | Wykrywanie patogenów, badania genów lekooporności, ocena skuteczności leków, diagnostyka chorób genetycznych. |
| latarnia molekularna | Wysoka specyficzność, fluorescencja, niskie tło | Cena jest wysoka, nadaje się tylko do określonego celu, projekt jest trudny, a cena jest wysoka. | Specyficzna analiza genów, analiza SNP |
2. Etapy eksperymentalne
2.1 O eksperymentalnym grupowaniu- w grupie musi być wiele studzienek i muszą występować powtórzenia biologiczne.
| ① | Pusta kontrola | Służy do wykrywania stanu wzrostu komórek w eksperymentach |
| ② | Kontrola negatywna siRNA (niespecyficzna sekwencja siRNA) | Wykazać specyfikę działania RNAi.siRNA może indukować niespecyficzną odpowiedź stresową w stężeniu 200 nM. |
| ③ | Kontrola odczynnika do transfekcji | Wykluczenie toksyczności odczynnika do transfekcji dla komórek lub wpływu na ekspresję docelowego genu |
| ④ | siRNA przeciwko genowi docelowemu | Znokautuj ekspresję docelowego genu |
| ⑤ (opcjonalnie) | pozytywne siRNA | Służy do rozwiązywania eksperymentalnych problemów systemowych i operacyjnych |
| ⑥ (opcjonalnie) | Fluorescencyjny kontrolny siRNA | Efektywność transfekcji komórek można obserwować pod mikroskopem |
2.2 Zasady projektowania podkładów
| Wzmocniony rozmiar fragmentu | Korzystnie 100-150 pz |
| Długość podkładu | 18-25 pb |
| zawartość CG | 30%-70%, najlepiej 45%-55% |
| Wartość Tm | 58-60℃ |
| Sekwencja | Unikaj ciągłego T/C;A/G ciągły |
| 3 sekwencja końcowa | Unikaj produktów bogatych w GC lub bogatych w AT;podstawą terminala jest korzystnie G lub C;najlepiej unikać T |
| Komplementarność | Unikaj komplementarnych sekwencji więcej niż 3 zasad w starterze lub między dwoma starterami |
| Specyficzność | Użyj wyszukiwania wybuchowego, aby potwierdzić specyficzność startera |
①SiRNA jest specyficzny dla gatunku, a sekwencje różnych gatunków będą różne.
②SiRNA jest pakowany w liofilizowany proszek, który można stabilnie przechowywać przez 2-4 tygodnie w temperaturze pokojowej.
2.3 Narzędzia lub odczynniki, które należy wcześniej przygotować
| Podkład (odniesienie wewnętrzne) | W tym dwa do przodu i do tyłu |
| Startery (gen docelowy) | W tym dwa do przodu i do tyłu |
| Docelowy Si RNA (3 paski) | Generalnie firma zsyntetyzuje 3 paski, a następnie wybierze jeden z trzech metodą RT-PCR |
| Zestaw do transfekcji | Lipo2000 itp. |
| Zestaw do szybkiej ekstrakcji RNA | Do ekstrakcji RNA po transfekcji |
| Zestaw do szybkiej odwrotnej transkrypcji | do syntezy cDNA |
| Zestaw do amplifikacji PCR | 2×Super SYBR Zielony qPCR Master Mix |
2.4 Jeśli chodzi o kwestie, na które należy zwrócić uwagę na poszczególnych etapach eksperymentu:
① Proces transfekcji siRNA
1. Do posiewu można wybrać płytkę 24-dołkową, 12-dołkową lub 6-dołkową (średnie stężenie RNA proponowane w każdym dołku 24-dołkowej płytki wynosi około 100-300 ng/ul), a optymalna gęstość transfekcji komórek wynosi około 60%-80%
2. Etapy transfekcji i specyficzne wymagania są ściśle zgodne z instrukcjami.
3. Po transfekcji próbki można pobrać w ciągu 24-72 godzin w celu wykrycia mRNA (RT-PCR) lub wykrycia białka w ciągu 48-96 godzin (WB)
② Proces ekstrakcji RNA
1. Zapobiegaj zanieczyszczeniu egzogennymi enzymami.Obejmuje to głównie ścisłe noszenie masek i rękawiczek;używanie wysterylizowanych końcówek do pipet i probówek EP;woda użyta w eksperymencie musi być wolna od RNaz.
2. Zaleca się wykonanie dwukrotnie czynności sugerowanych w zestawie do szybkiej ekstrakcji, co naprawdę poprawi czystość i wydajność.
3. Ciecz odpadowa nie może dotykać kolumny RNA.
③ Kwantyfikacja RNA
Po ekstrakcji RNA można go określić ilościowo bezpośrednio za pomocą Nanodrop, a minimalny odczyt może wynosić nawet 10 ng/ul.
④Proces odwrotnej transkrypcji
1. Ze względu na wysoką czułość RT-qPCR należy wykonać co najmniej 3 równoległe dołki dla każdej próbki, aby kolejne Ct nie były zbyt różne lub SD nie było zbyt duże do analizy statystycznej.
2. Nie zamrażaj i nie rozmrażaj Master mix wielokrotnie.
3. Każdą rurkę/otwór należy wymienić na nową końcówkę!Nie używaj ciągle tej samej końcówki pipety do dodawania próbek!
4. Film przyczepiony do płytki 96-dołkowej po dodaniu próbki należy wygładzić płytką.Najlepiej odwirować przed włożeniem do maszyny, aby płyn na ściankach probówki mógł spłynąć i usunąć pęcherzyki powietrza.
⑤Wspólna analiza krzywej
| Brak okresu wzrostu logarytmicznego | Możliwe wysokie stężenie matrycy |
| Brak wartości CT | Nieprawidłowe kroki wykrywania sygnałów fluorescencyjnych; degradacja starterów lub sond – jej integralność można wykryć za pomocą elektroforezy PAGE; niewystarczająca ilość szablonu; degradacja matryc – unikanie wprowadzania zanieczyszczeń oraz wielokrotnego zamrażania i rozmrażania w przygotowaniu próbek; |
| Ct>38 | Niska wydajność wzmocnienia;Produkt PCR jest za długi;różne składniki reakcji ulegają degradacji |
| Liniowa krzywa wzmocnienia | Sondy mogą ulec częściowej degradacji w wyniku powtarzających się cykli zamrażania i rozmrażania lub długotrwałej ekspozycji na światło |
| Różnica w podwójnych otworach jest szczególnie duża | Roztwór reakcyjny nie jest całkowicie stopiony lub roztwór reakcyjny nie jest wymieszany;łaźnia termiczna aparatu PCR jest zanieczyszczona substancjami fluorescencyjnymi |
2.5 O analizie danych
Analizę danych qPCR można podzielić na kwantyfikację względną i kwantyfikację bezwzględną.Na przykład komórki w grupie leczonej w porównaniu z komórkami w grupie kontrolnej,
Ile razy zmienia się mRNA genu X, jest to względna ocena ilościowa;w pewnej liczbie komórek mRNA genu X
Ile jest kopii, to absolutna kwantyfikacja.Zwykle w laboratorium najczęściej stosujemy względną metodę ilościową.Zazwyczaj,metoda 2-ΔΔctjest najczęściej używany w eksperymentach, więc tylko ta metoda zostanie tutaj szczegółowo omówiona.
Metoda 2-ΔΔct: Otrzymany wynik jest różnicą w ekspresji genu docelowego w grupie eksperymentalnej w stosunku do genu docelowego w grupie kontrolnej.Wymagane jest, aby wydajność amplifikacji zarówno genu docelowego, jak i wewnętrznego genu referencyjnego była bliska 100%, a względne odchylenie nie powinno przekraczać 5%.
Metoda obliczania jest następująca:
Δct grupa kontrolna = wartość ct genu docelowego w grupie kontrolnej – wartość ct wewnętrznego genu referencyjnego w grupie kontrolnej
Δct grupa eksperymentalna = wartość ct genu docelowego w grupie eksperymentalnej – wartość ct wewnętrznego genu referencyjnego w grupie eksperymentalnej
ΔΔct=Δct grupa eksperymentalna – Δct grupa kontrolna
Na koniec oblicz wielokrotność różnicy poziomów ekspresji:
Change Fold=2-ΔΔct (funkcja Excela odpowiada POWER)
Produkty powiązane:
Czas postu: 20-05-2023