Ⅰ. Zwiększ czułość układu reakcji:
1. Oddzielne wysokiej jakości RNA:
Udana synteza cDNA pochodzi z wysokiej jakości RNA.Wysokiej jakości RNA powinno zapewniać co najmniej łączną dłuższą żywotność i nie zawiera inhibitorów, które nie zawierają enzymów rejestrujących, takich jak EDTA czy SDS.Jakość RNA określa maksymalną wartość informacji o sekwencji, którą można przepisać na cDNA.Ogólna metoda oczyszczania RNA jest metodą etapową z użyciem izocyjanianu/kwasydofenolu.Aby zapobiec zanieczyszczeniu RNazą, RNA oddzielony od próbki bogatej w RNazę (takiej jak trzustka) wymaga przechowywania formaldehydu w celu zachowania wysokiej jakości RNA, co jest tym bardziej konieczne w przypadku przechowywania długoterminowego.RNA wyekstrahowany z wątroby szczura uległ zasadniczo degradacji po tygodniu przechowywania w wodzie, podczas gdy RNA wyekstrahowany ze śledziony szczura pozostał stabilny po trzech latach przechowywania w wodzie.Ponadto transkrypty większe niż 4 kb są bardziej wrażliwe na degradację śladowych RNaz niż małe transkrypty.W celu zwiększenia stabilności przechowywanej próbki RNA, RNA można rozpuścić w jonowej metaloaminie i przechowywać w temperaturze -70°C.Tylid użyty do zachowania RNA nie może zawierać innego obiektu, który degraduje RNA.RNA, które pochodzi z trzustki, można przechowywać w metalaminie przez co najmniej rok.Gdy jesteś gotowy do użycia RNA, możesz użyć następujących metod wytrącenia RNA: dodaj NaCl do 0,2 m i 4-krotności objętości etanolu, umieść w temperaturze pokojowej na 3-5 minut i odwiruj 10 000 × g przez 5 minut.
2. Użyj odwrotnej transkryptazy bez aktywności RNazyH (RNazyH-):
Inhibitory RNazy są często dodawane do reakcji odwrotnej transkrypcji w celu zwiększenia długości i wydajności syntezy cDNA.Inhibitor RNazy jest dodawany w pierwszej reakcji syntezy łańcuchowej w obecności buforów i czynników redukujących, takich jak DTT, ponieważ proces syntezy pre-cDNA denaturuje inhibitor, uwalniając w ten sposób związane RNazy, które degradują RNA.Inhibitor RNaz białkowych zapobiega jedynie degradacji RNA przez RNazy A, B, C i nie zapobiega RNazom na skórze, dlatego należy uważać, aby nie wprowadzać RNaz z palców pomimo stosowania tych inhibitorów.
Odwrotna transkryptaza katalizuje konwersję RNA do cDNA.Zarówno M-MLV, jak i AMV mają endogenną aktywność RNazyH oprócz własnej aktywności polimerazy.Aktywność RNazy H konkuruje z aktywnością polimerazy o heterozygotyczne nici utworzone między matrycami RNA a starterami DNA lub niciami przedłużającymi cDNA i degraduje RNA: nici RNA w kompleksach DNA.Matryce RNA zdegradowane przez aktywność RNazyH nie mogą być już stosowane jako skuteczne substraty do syntezy cDNA, zmniejszając wydajność i długość syntezy cDNA.Zatem wyeliminowanie lub znaczne zmniejszenie aktywności RNazy H odwrotnej transkryptazy byłoby bardzo korzystne.
Odwrotna transkryptaza SuperScriptⅡ, odwrotna transkryptaza MMLV RNazy H- i odwrotna transkryptaza thermoScript, AMV RNazy H- dały cDNA pełnej długości niż MMLV i AMV.Na czułość RT-PCR ma wpływ ilość zsyntetyzowanego cDNA.ThermoScript jest znacznie bardziej czuły niż AMV.Wielkość produktów RT-PCR jest ograniczona przez zdolność odwrotnej transkryptazy do syntezy cDNA, zwłaszcza podczas klonowania większych Cdna.W porównaniu z MMLV, SuperScripⅡ znacznie zwiększył wydajność długich produktów RT-PCR.Odwrotna transkryptaza RNazyH- zwiększa również stabilność termiczną, dzięki czemu reakcję można prowadzić w temperaturach wyższych niż normalnie 37-42℃.W sugerowanych warunkach syntezy zastosowano startery oligo(dT) i 10 μCi [alfa-p]dCTP.Całkowitą produkcję pierwszego łańcucha obliczono metodą wytrącania TCA.Pełnej długości cDNA analizowano stosując usuwanie pasków sortowanych pod względem wielkości i zliczanie w alkalicznym żelu agarozowym.
3. Zwiększ temperaturę zachowania ciepła odwrotnej transkrypcji:
Wyższa temperatura przetrzymywania pomaga otworzyć drugorzędową strukturę RNA i zwiększyć wydajność reakcji.W przypadku większości matryc RNA trzymanie RNA i startera w temperaturze 65°C bez buforu lub soli, a następnie szybkie schłodzenie ich na lodzie eliminuje większość struktur drugorzędowych i umożliwia związanie starterów.Jednak niektóre szablony nadal mają strukturę drugorzędową, nawet po denaturacji termicznej.Amplifikację tych trudnych matryc można przeprowadzić przy użyciu odwrotnej transkryptazy ThermoScript i umieszczając reakcję odwrotnej transkryptazy w wyższych temperaturach w celu poprawy amplifikacji.Wyższe temperatury przetrzymywania mogą również zwiększyć specyficzność, zwłaszcza gdy synteza cDNA jest przeprowadzana przy użyciu starterów specyficznych dla genu (GSPS) (patrz rozdział 3).Jeśli używasz GSP, upewnij się, że wartość Tm podkładu jest taka sama jak oczekiwana temperatura przetrzymywania.Nie stosować starterów oligo(dT) i losowych powyżej 60 ℃.Losowe podkłady należy trzymać w temperaturze 25 ℃ przez 10 minut przed zwiększeniem do 60 ℃.Oprócz stosowania wyższych temperatur odwrotnej transkrypcji, specyficzność można poprawić poprzez bezpośrednie przeniesienie mieszaniny RNA/starter z temperatury denaturacji 65℃ do temperatury utrzymywania odwrotnej transkrypcji i dodanie wstępnie ogrzanej 2× mieszaniny reakcyjnej (synteza inicjacji termicznej cDNA).Takie podejście pomaga zapobiegać parowaniu zasad międzycząsteczkowych, które występuje w niższych temperaturach.Używanie aparatu do PCR upraszcza wiele przełączników temperatury wymaganych do RT-PCR.
Termostabilizowana polimeraza Tth działa jak polimeraza DNA w obecności Mg2+ i polimeraza RNA w obecności Mn2+.Może utrzymywać ciepło do 65 ℃.Jednak obecność Mn2+ podczas PCR zmniejsza wierność, co sprawia, że polimeraza Tth jest mniej odpowiednia do amplifikacji o wysokiej precyzji, takiej jak klonowanie cDNA.Ponadto Tth jest mniej wydajny w odwrotnej transkrypcji, co zmniejsza czułość, a ponieważ pojedynczy enzym może przeprowadzić odwrotną transkrypcję i PCR, reakcje kontrolne bez odwrotnej transkrypcji nie mogą być użyte do odróżnienia zamplifikowanych produktów cDNA od zanieczyszczonego genomowego DNA.
4. Dodatek promujący odwrotną transkrypcję:
Dodanie dodatków, w tym gliceryny i DMSO, do pierwszej reakcji syntezy łańcucha może zmniejszyć stabilność podwójnej nici kwasu nukleinowego i odwinąć drugorzędową strukturę RNA.Można dodać do 20% gliceryny lub 10% DMSO bez wpływu na aktywność SuperScriptⅡ lub MMLV.AMV może również tolerować do 20% glicerolu bez zmniejszania aktywności.Aby zmaksymalizować czułość RT-PCR w reakcji odwrotnej transkrypcji SuperScriptⅡ, można dodać 10% glicerolu i izolować w temperaturze 45℃.Jeśli do PCR doda się 1/10 produktu reakcji retrotranskrypcji, stężenie glicerolu w reakcji amplifikacji wynosi 0,4%, co nie wystarcza do zahamowania reakcji PCR.
5. Przetwarzanie RNazy:
Czułość można poprawić, traktując reakcje syntezy cDNA RNaząH przed PCR.W przypadku niektórych matryc uważa się, że RNA w reakcji syntezy cDNA zapobiega wiązaniu zamplifikowanych produktów, w którym to przypadku traktowanie RNaząH może zwiększyć czułość.Ogólnie, obróbka RNaząH jest wymagana do amplifikacji stosunkowo długiej, pełnej długości docelowej matrycy cDNA, takiej jak scheroza guzowataⅡ z niską liczbą kopii.W przypadku tego trudnego szablonu RNazaH wzmocniła sygnał generowany przez cDNA zsyntetyzowany przez SuperScriptⅡ lub AMV.W przypadku większości reakcji RT-PCR obróbka RNaząH jest opcjonalna, ponieważ etap denaturacji PCR w izolacji 95 ℃ zazwyczaj hydrolizuje RNA z kompleksu RNA: DNA.
6. Udoskonalone metody wykrywania małych ilości RNA:
RT-PCR jest szczególnie trudny, gdy dostępne są tylko niewielkie ilości RNA.Dodatek glikogenu jako nośnika podczas rozdzielania RNA pomaga zwiększyć wydajność małych próbek.Równocześnie z Trizolem można dodać glikogen wolny od RNaz.Glikogen jest rozpuszczalny w wodzie i może pozostać w fazie wodnej z RNA, aby pomóc w późniejszym wytrącaniu.Zalecane stężenie glikogenu wolnego od RNaz wynosi 250 μg/ml dla próbek mniejszych niż 50 mg tkanki lub 106 hodowanych komórek.
Dodatek acetylowanego BSA do reakcji odwrotnej transkrypcji przy użyciu SuperScriptⅡ może zwiększyć czułość, a dla małych ilości RNA zmniejszenie ilości SuperScriptⅡ i dodanie 40 jednostek inhibitora nukleazy RnaseOut może poprawić poziom detekcji.Jeśli glikogen jest używany do rozdzielania RNA, nadal zaleca się dodanie inhibitorów BSA lub RNazy w celu odwrócenia reakcji transkrypcji przy użyciu SuperScriptⅡ.
Ⅱ. Zwiększ specyficzność RT-PCR
1. Synteza cNDA:
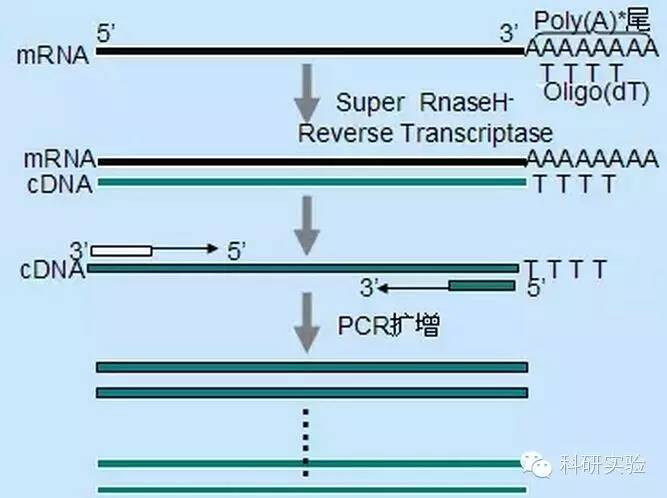
Do zainicjowania syntezy pierwszej nici cDNA można zastosować trzy różne metody, a względna specyficzność każdej metody wpływa na ilość i rodzaj syntetyzowanego cDNA.
Metoda losowego startera jest najmniej specyficzna z trzech metod.Startery są przyłączane w wielu miejscach w całym transkrypcie, aby wytworzyć cDNA o krótkiej, częściowej długości.Ta metoda jest często stosowana do uzyskiwania sekwencji końcowych 5' i cDNA z matryc RNA z drugorzędowymi regionami strukturalnymi lub miejscami końcowymi, których odwrotna transkryptaza nie może replikować.Aby uzyskać najdłuższy cDNA, stosunek starterów do RNA w każdej próbce RNA należy określić empirycznie.Początkowe stężenie losowych starterów mieści się w zakresie od 50 do 250 ng na 20 μl układu reakcyjnego.Ponieważ cDNA zsyntetyzowany z całkowitego RNA przy użyciu losowych starterów to głównie rybosomalny RNA, poli(A)+RNA jest na ogół wybierany jako matryca.
Inicjacja oligo(dT) jest bardziej specyficzna niż losowe startery.Hybrydyzuje z ogonem poli(A) znajdującym się na końcu 3' mRNA w większości komórek eukariotycznych.Ponieważ poli(A)+RNA stanowi około 1% do 2% całkowitego RNA, ilość i złożoność cDNA jest znacznie mniejsza niż w przypadku użycia losowych starterów.Ze względu na swoją wysoką specyficzność, oligo(dT) na ogół nie wymaga optymalizacji stosunku RNA do startera i selekcji poli(A)+.Zaleca się stosowanie 0,5 μg oligo(dT) na 20 μl układu reakcyjnego.oligo(dT)12-18 jest odpowiedni dla większości RT-PCR.System ThermoScript RT-PCR zapewnia oligo(dT)20 ze względu na dobrą stabilność termiczną i nadaje się do wyższych temperatur przetrzymywania.
Primery specyficzne dla genu (GSP) są najlepszymi starterami specyficznymi dla etapu odwrotnej transkrypcji.GSP jest antysensownym oligonukleozydem, który może specyficznie hybrydyzować z docelowymi sekwencjami RNA, zamiast łączyć wszystkie Rna, takie jak losowe startery lub oligo(dT).Zasady stosowane do projektowania starterów PCR mają również zastosowanie do projektowania reakcji odwrotnej transkrypcji GSP.GSP może być tą samą sekwencją, co starter do amplifikacji połączony na końcu mRNA3' lub GSP można zaprojektować tak, aby był połączony w dół ze starterem do odwrotnej amplifikacji.W przypadku niektórych amplifikowanych obiektów konieczne jest zaprojektowanie więcej niż jednego antysensownego startera do skutecznego RT-PCR, ponieważ drugorzędowa struktura docelowego RNA może uniemożliwić wiązanie startera.Sugeruje się użycie 1 pmol antysensownego GSP w pierwszym układzie reakcji syntezy łańcuchowej 20 µl.
2. Zwiększ temperaturę zachowania ciepła odwrotnej transkrypcji:
Aby w pełni wykorzystać specyficzność GSP, należy zastosować odwrotną transkryptazę o wysokiej stabilności termicznej.Termostabilną odwrotną transkryptazę można izolować w wyższych temperaturach, aby zwiększyć rygor reakcji.Na przykład, jeśli GSP jest hybrydyzowany w temperaturze 55°C, specyficzność GSP nie jest w pełni wykorzystywana, jeśli odwrotną transkrypcję przeprowadza się w temperaturze 37°C z niskim rygorem przy użyciu AMV lub M-MLV.Jednak SuperScripⅡ i ThermoScript mogą reagować w temperaturze 50℃ lub wyższej, co eliminuje niespecyficzne produkty wytwarzane w niższych temperaturach.W celu uzyskania maksymalnej specyficzności mieszaninę RNA/starter można przenieść bezpośrednio z temperatury denaturacji 65℃ do temperatury utrzymywania odwrotnej transkrypcji z dodatkiem podgrzanej 2 x mieszaniny reakcyjnej (termiczna inicjacja syntezy cDNA).Pomaga to zapobiegać parowaniu zasad między cząsteczkami w niskich temperaturach.Używanie aparatu do PCR upraszcza wiele zmian temperatury wymaganych do RT-PCR.
3. Zmniejsz zanieczyszczenie genomowym DNA:
Jedną z potencjalnych trudności związanych z RT-PCR jest to, że RNA zanieczyszcza genomowy DNA.Zastosowanie lepszych metod separacji RNA, takich jak Trizol Reagent, zmniejsza zanieczyszczenie genomowym DNA w preparatach RNA.Aby uniknąć produktów wytwarzanych z genomowego DNA, RNA można potraktować DnasⅠ stopnia amplifikacji w celu usunięcia zanieczyszczonego DNA przed odwrotną transkrypcją.Próbki trzymano w temperaturze 65°C w 2,0 mM EDTA przez 10 minut w celu zakończenia trawienia DNaząⅠ.EDTA chelatuje jony magnezu, aby zapobiec zależnej od jonów magnezu hydrolizie RNA, która zachodzi w wysokich temperaturach.
W celu oddzielenia zamplifikowanego cDNA od produktu amplifikacji genomowego DNA można zaprojektować startery, które hybrydyzują oddzielnie z oddzielonym eksonem.Produkty PCR pochodzące z cDNA będą krótsze niż te pochodzące z zanieczyszczonego genomowego DNA.Na każdej matrycy RNA przeprowadza się również kontrolowany eksperyment bez odwrotnej transkrypcji, aby określić, czy dany fragment pochodzi z genomowego DNA, czy z cDNA.Produkty PCR otrzymane przy braku odwrotnej transkrypcji pochodzą z genomu.
Powiązany produkt
- Zestaw jednoetapowy umożliwia przeprowadzenie odwrotnej transkrypcji i PCR w tej samej probówce.Wystarczy dodać matrycowy RNA, specyficzne startery PCR i wolne od RNazy ddH2O.
-Analiza ilościowa RNA w czasie rzeczywistym może być przeprowadzona szybko i dokładnie.
-Zestaw wykorzystuje unikalny odczynnik do odwrotnej transkrypcji Foregene i polimerazę Foregene HotStar Taq DNA w połączeniu z unikalnym systemem reakcji, aby skutecznie poprawić wydajność amplifikacji i specyficzność reakcji.
-Zoptymalizowany system reakcji sprawia, że reakcja ma wyższą czułość wykrywania, większą stabilność termiczną i lepszą tolerancję.
-Wydajna zdolność do usuwania gDNA, która może usunąć gDNA z szablonu w ciągu 2 minut.
-Wydajny system odwrotnej transkrypcji, synteza pierwszej nici cDNA zajmuje tylko 15 minut.
-Złożone szablony: szablony o wysokiej zawartości GC i złożonej strukturze drugorzędowej można również odwracać z dużą wydajnością.
- System odwrotnej transkrypcji o wysokiej czułości, szablony na poziomie pg mogą również uzyskać wysokiej jakości cDNA.
- System odwrotnej transkrypcji ma wysoką stabilność termiczną, optymalna temperatura reakcji wynosi 42 ℃ i nadal ma dobrą wydajność odwrotnej transkrypcji przy 50 ℃.
Czas postu: 07-03-2023