一、Zwiększ czułość układu reakcji:
1. Izoluj wysokiej jakości RNA:
Udana synteza cDNA pochodzi z wysokiej jakości RNA.Wysokiej jakości RNA powinno być co najmniej pełnej długości i wolne od inhibitorów odwrotnej transkryptazy, takich jak EDTA lub SDS.Jakość RNA określa maksymalną ilość informacji o sekwencji, którą można przepisać na cDNA.Powszechną metodą oczyszczania RNA jest metoda jednoetapowa z użyciem izotiocyjanianu guanidyny/kwaśnego fenolu.Aby zapobiec zanieczyszczeniu śladowymi ilościami RNazy, RNA wyizolowany z próbek bogatych w RNazy (takich jak trzustka) musi być przechowywany w formaldehydzie w celu zachowania wysokiej jakości RNA, zwłaszcza w przypadku długotrwałego przechowywania.RNA wyekstrahowane z wątroby szczura uległo zasadniczo degradacji po przechowywaniu w wodzie przez jeden tydzień, podczas gdy RNA wyekstrahowane ze śledziony szczura pozostawało stabilne po przechowywaniu w wodzie przez 3 lata.Ponadto transkrypty dłuższe niż 4 kb są bardziej wrażliwe na degradację przez śladowe RNazy niż małe transkrypty.Aby zwiększyć stabilność przechowywanych próbek RNA, RNA można rozpuścić w dejonizowanym formamidzie i przechowywać w temperaturze -70°C.Formamid używany do konserwacji RNA musi być wolny od resztek degradujących RNA.RNA z trzustki można przechowywać w formamidzie przez co najmniej rok.Przygotowując się do użycia RNA, można zastosować następującą metodę wytrącenia RNA: dodać NaCl do 0,2 M i 4-krotności objętości etanolu, umieścić w temperaturze pokojowej na 3-5 minut i wirować przy 10 000 x g przez 5 minut.
2. Użyj odwrotnej transkryptazy RNazyH-nieaktywnej (RNazyH-):
Inhibitory RNazy są często dodawane do reakcji odwrotnej transkrypcji w celu zwiększenia długości i wydajności syntezy cDNA.Inhibitory RNazy należy dodać podczas reakcji syntezy pierwszej nici w obecności buforu i czynnika redukującego (takiego jak DTT), ponieważ proces przed syntezą cDNA denaturuje inhibitor, uwalniając w ten sposób związaną RNazę, która może degradować RNA.Inhibitory RNaz białkowych zapobiegają jedynie degradacji RNA przez RNazy A, B, C i nie zapobiegają RNazy na skórze, dlatego należy uważać, aby nie wprowadzać RNazy z palców pomimo stosowania tych inhibitorów.
Odwrotna transkryptaza katalizuje konwersję RNA do cDNA.Zarówno M-MLV, jak i AMV mają endogenną aktywność RNazyH oprócz własnej aktywności polimerazy.Aktywność RNazyH i aktywność polimerazy konkurują ze sobą o nić hybrydową utworzoną między matrycą RNA a starterem DNA lub nicią przedłużającą cDNA i degradują nić RNA w kompleksie RNA:DNA.Matryca RNA zdegradowana przez aktywność RNazy H nie może już służyć jako efektywny substrat do syntezy cDNA, co zmniejsza wydajność i długość syntezy cDNA.Dlatego korzystne byłoby wyeliminowanie lub znaczne zmniejszenie aktywności RNazy H odwrotnej transkryptazy.
Odwrotna transkryptaza SuperScript Ⅱ, odwrotna transkryptaza RNazyH-MMLV i odwrotna transkryptaza thermoScript, RNazaH-AMV, mogą uzyskać więcej cDNA pełnej długości niż MMLV i AMV.Na czułość RT-PCR będzie miała wpływ ilość syntezy cDNA.ThermoScript jest znacznie bardziej czuły niż AMV.Wielkość produktów RT-PCR jest ograniczona przez zdolność odwrotnej transkryptazy do syntezy cDNA, zwłaszcza podczas klonowania większych cDNA.W porównaniu z MMLV, SuperScripⅡ znacznie zwiększył wydajność długich produktów RT-PCR.Odwrotna transkryptaza RNazy H ma również zwiększoną termostabilność, dzięki czemu reakcję można prowadzić w temperaturach wyższych niż normalne 37-42°C.W sugerowanych warunkach syntezy użyj startera oligo(dT) i 10 μCi [α-P]dCTP.Całkowity uzysk pierwszej nici obliczono metodą wytrącania TCA.Pełnej długości cDNA analizowano stosując posortowane pod względem wielkości prążki wycięte i zliczone na alkalicznym żelu agarozowym.
3. Podnieś temperaturę inkubacji dla odwrotnej transkrypcji:
Wyższa temperatura inkubacji pomaga otworzyć drugorzędową strukturę RNA, zwiększając wydajność reakcji.W przypadku większości matryc RNA inkubacja RNA i starterów w temperaturze 65°C bez buforu lub soli, a następnie szybkie schłodzenie na lodzie wyeliminuje większość struktur drugorzędowych i umożliwi związanie starterów.Jednak niektóre szablony nadal mają struktury drugorzędowe, nawet po denaturacji termicznej.Amplifikację tych trudnych matryc można przeprowadzić przy użyciu odwrotnej transkryptazy ThermoScript i umieszczenia reakcji odwrotnej transkrypcji w wyższej temperaturze w celu poprawy amplifikacji.Wyższa temperatura inkubacji może również zwiększyć specyficzność, zwłaszcza gdy do syntezy cDNA stosuje się startery specyficzne dla genu (GSP) (patrz rozdział 3).Jeśli używasz GSP, upewnij się, że Tm starterów jest taka sama jak oczekiwana temperatura inkubacji.Nie używać starterów oligo(dT) i losowych w temperaturze powyżej 60°C.Losowe startery wymagają inkubacji w temperaturze 25°C przez 10 minut przed zwiększeniem do 60°C.Oprócz zastosowania wyższej temperatury odwrotnej transkrypcji, specyficzność można również poprawić poprzez bezpośrednie przeniesienie mieszaniny RNA/starter z temperatury denaturacji 65°C do temperatury inkubacji odwrotnej transkrypcji i dodanie wstępnie ogrzanej 2x mieszaniny reakcyjnej (synteza cDNA na gorąco).Takie podejście pomaga zapobiegać parowaniu zasad międzycząsteczkowych, które występuje w niższych temperaturach.Wielokrotne przełączanie temperatury wymagane do RT-PCR można uprościć, stosując termocykler.
Termostabilna polimeraza Tth działa jak polimeraza DNA w obecności Mg2+ i jako polimeraza RNA w obecności Mn2+.Może być utrzymywany w cieple w maksymalnej temperaturze 65°C.Jednak obecność Mn2+ podczas PCR zmniejsza wierność, co sprawia, że polimeraza Tth jest mniej odpowiednia do bardzo precyzyjnych amplifikacji, takich jak klonowanie cDNA.Ponadto Tth ma niską wydajność odwrotnej transkrypcji, co zmniejsza czułość, a ponieważ odwrotną transkrypcję i PCR można przeprowadzić za pomocą jednego enzymu, reakcje kontrolne bez odwrotnej transkrypcji nie mogą być stosowane do porównywania produktów amplifikacji cDNA z zanieczyszczającym genomowym DNA.Produkty amplifikacji rozdzielono.
4. Dodatki promujące odwrotną transkrypcję:
Dodatki, w tym glicerol i DMSO, są dodawane do reakcji syntezy pierwszej nici, co może zmniejszyć stabilność dwuniciowego kwasu nukleinowego i rozwiązać drugorzędową strukturę RNA.Można dodać do 20% glicerolu lub 10% DMSO bez wpływu na aktywność SuperScript II lub MMLV.AMV może również tolerować do 20% glicerolu bez utraty aktywności.Aby zmaksymalizować czułość RT-PCR w reakcji odwrotnej transkrypcji SuperScriptⅡ, można dodać 10% glicerolu i inkubować w temperaturze 45°C.Jeśli do PCR doda się 1/10 produktu reakcji odwrotnej transkrypcji, to stężenie glicerolu w reakcji amplifikacji wynosi 0,4%, co nie wystarcza do zahamowania PCR.
5. Leczenie RNazą:
Traktowanie reakcji syntezy cDNA RNaząH przed PCR może zwiększyć czułość.W przypadku niektórych matryc uważa się, że RNA w reakcji syntezy cDNA zapobiega wiązaniu produktów amplifikacji, w którym to przypadku traktowanie RNaząH może zwiększyć czułość.Ogólnie, traktowanie RNaząH jest konieczne przy amplifikacji dłuższych, pełnej długości docelowych matryc cDNA, takich jak scheroza guzowata II o niskiej liczbie kopii.W przypadku tego trudnego szablonu traktowanie RNaząH wzmocniło sygnał wytwarzany przez SuperScript II lub cDNA zsyntetyzowany przez AMV.W przypadku większości reakcji RT-PCR traktowanie RNaząH jest opcjonalne, ponieważ etap denaturacji PCR w temperaturze 95°C generalnie hydrolizuje RNA w kompleksie RNA:DNA.
6. Ulepszenie metody wykrywania małych RNA:
RT-PCR jest szczególnie trudny, gdy dostępne są tylko niewielkie ilości RNA.Glikogen dodany jako nośnik podczas izolacji RNA pomaga zwiększyć wydajność małych próbek.Glikogen wolny od RNaz można dodać w tym samym czasie co Trizol.Glikogen jest rozpuszczalny w wodzie i może być utrzymywany w fazie wodnej z RNA, aby wspomóc późniejsze wytrącanie.W przypadku próbek mniejszych niż 50 mg tkanki lub 106 hodowanych komórek zalecane stężenie glikogenu wolnego od RNaz wynosi 250 μg/ml.
Dodanie acetylowanego BSA do reakcji odwrotnej transkrypcji przy użyciu SuperScript II może zwiększyć czułość, a dla małych ilości RNA zmniejszenie ilości SuperScript II i dodanie 40 jednostek inhibitora nukleazy RNaseOut może zwiększyć poziom wykrywania.Jeśli glikogen jest używany w procesie izolacji RNA, nadal zaleca się dodanie BSA lub inhibitora RNazy przy użyciu SuperScript II do reakcji odwrotnej transkrypcji.
二, Zwiększ specyficzność RT-PCR
1. Synteza CND:
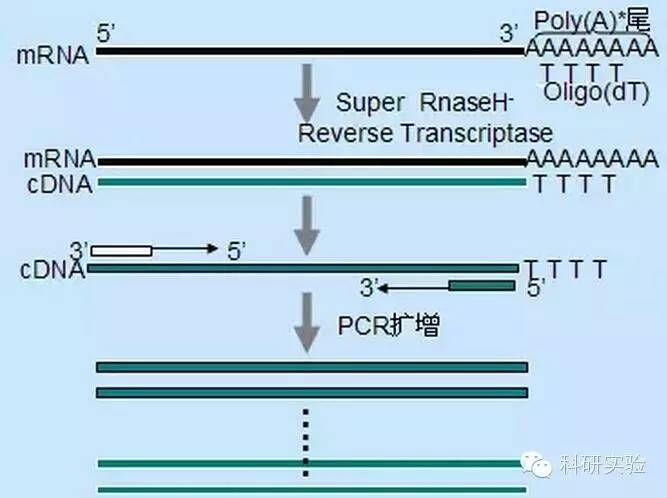
Syntezę pierwszej nici cDNA można zainicjować trzema różnymi metodami, których względna specyficzność wpływa na ilość i rodzaj syntetyzowanego cDNA.
Metoda losowego startera była najmniej specyficzna z trzech metod.Startery przyłączają się w wielu miejscach transkryptu, generując krótkie cDNA o częściowej długości.Metoda ta jest często wykorzystywana do uzyskiwania sekwencji końca 5' oraz do uzyskiwania cDNA z matryc RNA z regionami o strukturze drugorzędowej lub z miejscami terminacji, które nie mogą być replikowane przez odwrotną transkryptazę.Aby uzyskać najdłuższy cDNA, stosunek starterów do RNA w każdej próbce RNA należy określić empirycznie.Początkowe stężenie losowych starterów mieściło się w zakresie od 50 do 250 ng na 20 μl reakcji.Ponieważ cDNA zsyntetyzowany z całkowitego RNA przy użyciu losowych starterów jest głównie rybosomalnym RNA, poli(A)+RNA jest na ogół wybierany jako matryca.
Startery oligo(dT) są bardziej specyficzne niż startery losowe.Hybrydyzuje z ogonem poli(A) znajdującym się na końcu 3' większości eukariotycznych mRNA.Ponieważ poli(A)+ RNA stanowi około 1% do 2% całkowitego RNA, ilość i złożoność cDNA jest znacznie mniejsza niż w przypadku losowych starterów.Ze względu na swoją wysoką specyficzność, oligo(dT) na ogół nie wymaga optymalizacji stosunku RNA do starterów i selekcji poli(A)+.Zaleca się stosowanie 0,5 μg oligo(dT) na 20 μl układu reakcyjnego.oligo(dT)12-18 jest odpowiedni dla większości RT-PCR.System ThermoScript RT-PCR oferuje oligo(dT)20 ze względu na lepszą stabilność termiczną dla wyższych temperatur inkubacji.
Startery specyficzne dla genu (GSP) są najbardziej specyficznymi starterami dla etapu odwrotnej transkrypcji.GSP jest antysensownym oligonukleotydem, który może specyficznie hybrydyzować z docelową sekwencją RNA, w przeciwieństwie do losowych starterów lub oligo(dT), które przyłączają się do wszystkich RNA.Te same zasady stosowane do projektowania starterów PCR mają zastosowanie do projektowania GSP w reakcjach odwrotnej transkrypcji.GSP może być tą samą sekwencją co starter do amplifikacji, który przyłącza się do końca 3' mRNA, lub GSP można zaprojektować tak, aby przyłączał się poniżej startera do odwrotnej amplifikacji.W przypadku niektórych osobników poddanych amplifikacji należy zaprojektować więcej niż jeden antysensowny starter do skutecznego RT-PCR, ponieważ drugorzędowa struktura docelowego RNA może uniemożliwić wiązanie startera.Zaleca się stosowanie 1 pmol antysensownego GSP w 20 μl reakcji syntezy pierwszej nici.
2. Podnieś temperaturę inkubacji dla odwrotnej transkrypcji:
Aby w pełni wykorzystać zalety specyficzności GSP, należy zastosować odwrotną transkryptazę o wyższej termostabilności.Termostabilne odwrotne transkryptazy można inkubować w wyższych temperaturach, aby zwiększyć ostrość reakcji.Na przykład, jeśli GSP hybrydyzuje w temperaturze 55°C, specyficzność GSP nie zostanie w pełni wykorzystana, jeśli do odwrotnej transkrypcji zastosuje się AMV lub M-MLV w niskiej ostrości 37°C.Jednak SuperScript II i ThermoScript można poddawać reakcji w temperaturze 50°C lub wyższej, co wyeliminuje niespecyficzne produkty powstające w niższych temperaturach.Aby uzyskać maksymalną specyficzność, mieszaninę RNA/starterów można przenieść bezpośrednio z temperatury denaturacji 65°C do temperatury inkubacji z odwrotną transkrypcją i dodać do wstępnie ogrzanej mieszaniny reakcyjnej 2x (gorący start syntezy cDNA).Pomaga to zapobiegać parowaniu zasad międzycząsteczkowych w niskich temperaturach.Wielokrotne zmiany temperatury wymagane w RT-PCR można uprościć, stosując termocykler.
3. Zmniejsza zanieczyszczenie genomowym DNA:
Potencjalną trudnością napotkaną przy RT-PCR jest zanieczyszczenie genomowego DNA w RNA.Stosowanie dobrej metody izolacji RNA, takiej jak Trizol Reagent, zmniejszy ilość genomowego DNA zanieczyszczającego preparat RNA.Aby uniknąć produktów pochodzących z genomowego DNA, RNA można potraktować DNazą I stopnia do amplifikacji w celu usunięcia zanieczyszczającego DNA przed odwrotną transkrypcją.Trawienie DNazą I zakończono przez inkubację próbek w 2,0 mM EDTA przez 10 minut w temperaturze 65°C.EDTA może chelatować jony magnezu, zapobiegając zależnej od jonów magnezu hydrolizie RNA w wysokich temperaturach.
W celu oddzielenia zamplifikowanego cDNA od zanieczyszczających produktów amplifikacji genomowego DNA, można zaprojektować startery, które łączą się w oddzielne eksony.Produkty PCR pochodzące z cDNA będą krótsze niż te pochodzące z zanieczyszczonego genomowego DNA.Ponadto przeprowadzono eksperyment kontrolny bez odwrotnej transkrypcji na każdej matrycy RNA, aby określić, czy dany fragment pochodzi z genomowego DNA, czy z cDNA.Produkt PCR otrzymany bez odwrotnej transkrypcji pochodzi z genomu.
Czas postu: 16-05-2023








