PCR (reakcja łańcuchowa polimerazy) to jedna z technologii amplifikacji DNA in vitro, której historia sięga ponad 30 lat.
Technologię PCR zapoczątkował Kary Mullis z Cetus w USA w 1983 r. Mullis złożył wniosek o patent na PCR w 1985 r. iw tym samym roku opublikował pierwszy artykuł naukowy PCR na temat nauki.Mullis otrzymał Nagrodę Nobla w dziedzinie chemii w 1993 roku za swoją pracę.
Podstawowe zasady PCR
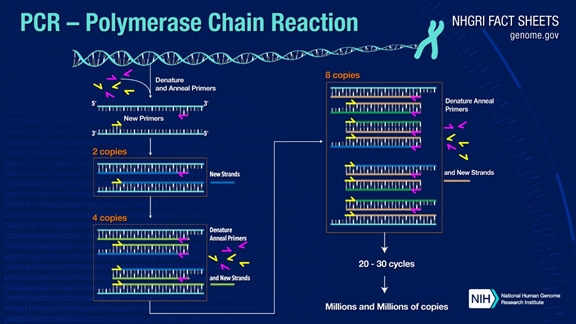
PCR może amplifikować docelowe fragmenty DNA ponad milion razy.Zasadą jest kataliza polimerazy DNA, przy użyciu rodzicielskiej nici DNA jako matrycy i specyficznego startera jako punktu wyjścia do wydłużania.Jest replikowany in vitro poprzez etapy takie jak denaturacja, hybrydyzacja i wydłużanie.Proces tworzenia nici potomnej komplementarnej do macierzystej nici matrycowej DNA.
Standardowy proces PCR dzieli się na trzy etapy:
1. Denaturacja: Użyj wysokiej temperatury, aby oddzielić podwójne nici DNA.Wiązanie wodorowe między podwójnymi łańcuchami DNA zostaje zerwane w wysokiej temperaturze (93-98℃).
2. Wygrzewanie: Po rozdzieleniu dwuniciowego DNA obniż temperaturę, aby starter mógł związać się z jednoniciowym DNA.
3. Wydłużenie: Polimeraza DNA zaczyna syntetyzować komplementarne nici wzdłuż nici DNA ze związanych starterów, gdy temperatura jest obniżona.Po zakończeniu wydłużania cykl się kończy, a liczba fragmentów DNA podwaja się
Powtarzając te trzy kroki 25-35 razy, liczba fragmentów DNA wzrośnie wykładniczo.
Pomysłowość PCR polega na tym, że można zaprojektować różne startery dla różnych genów docelowych, dzięki czemu fragmenty genów docelowych można powielić w krótkim czasie.
Jak dotąd PCR można podzielić na trzy kategorie, a mianowicie zwykły PCR, fluorescencyjny ilościowy PCR i cyfrowy PCR.
Pierwsza generacja zwykłego PCR
Użyj zwykłego instrumentu do amplifikacji PCR, aby powielić docelowy gen, a następnie użyj elektroforezy w żelu agarozowym, aby wykryć produkt, można przeprowadzić tylko analizę jakościową.
Główne wady PCR pierwszej generacji:
1. Skłonny do niespecyficznej amplifikacji i wyników fałszywie dodatnich.
2. Wykrywanie zajmuje dużo czasu, a operacja jest uciążliwa.
3. Można wykonać tylko test jakościowy
PCR w czasie rzeczywistym drugiej generacji
PCR w czasie rzeczywistym, znany również jako qPCR, wykorzystuje sondy fluorescencyjne, które mogą wskazywać postęp układu reakcyjnego i monitoruje akumulację zamplifikowanych produktów poprzez akumulację sygnałów fluorescencyjnych i ocenia wyniki na podstawie krzywej fluorescencji.Można to określić ilościowo za pomocą wartości Cq i krzywej wzorcowej.
Ponieważ technologia qPCR jest przeprowadzana w systemie zamkniętym, prawdopodobieństwo zanieczyszczenia jest zmniejszone, a sygnał fluorescencyjny można monitorować w celu detekcji ilościowej, dlatego jest ona najczęściej stosowana w praktyce klinicznej i stała się dominującą technologią w PCR.
Substancje fluorescencyjne stosowane w fluorescencyjnej ilościowej reakcji PCR w czasie rzeczywistym można podzielić na: sondę fluorescencyjną TaqMan, sygnalizatory molekularne oraz barwnik fluorescencyjny.
1) Sonda fluorescencyjna TaqMan:
Podczas amplifikacji PCR dodawana jest specyficzna sonda fluorescencyjna, podczas dodawania pary starterów.Sonda jest oligonukleotydem, a oba końce są oznaczone reporterową grupą fluorescencyjną i wygaszającą grupą fluorescencyjną.
Gdy sonda jest nienaruszona, sygnał fluorescencyjny emitowany przez grupę reporterową jest absorbowany przez grupę wygaszającą;podczas amplifikacji PCR aktywność egzonukleazy 5′-3′ enzymu Taq rozszczepia i degraduje sondę, tworząc reporterową grupę fluorescencyjną i wygaszacz Grupa fluorescencyjna jest oddzielana, dzięki czemu system monitorowania fluorescencji może odbierać sygnał fluorescencji, to znaczy za każdym razem, gdy nić DNA jest amplifikowana, tworzona jest cząsteczka fluorescencyjna, a gromadzenie się sygnału fluorescencyjnego jest całkowicie zsynchronizowane z tworzeniem produktu PCR.
2) barwnik fluorescencyjny SYBR:
W układzie reakcyjnym PCR dodaje się nadmiar barwnika fluorescencyjnego SYBR.Po niespecyficznym włączeniu barwnika fluorescencyjnego SYBR do podwójnej nici DNA, emituje on sygnał fluorescencyjny.Cząsteczka barwnika SYBR, która nie jest włączona do łańcucha, nie będzie emitować żadnego sygnału fluorescencyjnego, zapewniając w ten sposób sygnał fluorescencyjny. Wzrost produktów PCR jest całkowicie zsynchronizowany ze wzrostem produktów PCR.SYBR wiąże się tylko z dwuniciowym DNA, więc krzywą topnienia można wykorzystać do określenia, czy reakcja PCR jest specyficzna.
3) Latarnia molekularna:
Jest to podwójnie znakowana sonda oligonukleotydowa z pętlą łodygi, która tworzy strukturę spinki do włosów z około 8 zasadami na końcach 5 i 3.Sekwencje kwasów nukleinowych na obu końcach są komplementarnie sparowane, co powoduje, że grupa fluorescencyjna i grupa wygaszająca są szczelne.Zamknij, nie będzie wytwarzana żadna fluorescencja.
Po wytworzeniu produktu PCR, podczas procesu wyżarzania, środkowa część latarni molekularnej jest parowana z określoną sekwencją DNA, a gen fluorescencyjny jest oddzielany od genu wygaszacza w celu wytworzenia fluorescencji.
Główne wady PCR drugiej generacji:
Wciąż brakuje czułości, a wykrywanie próbek o niskiej liczbie kopii jest niedokładne.
Wpływ ma wartość tła, a wynik jest podatny na zakłócenia.
Gdy w układzie reakcyjnym znajdują się inhibitory PCR, wyniki wykrywania są podatne na zakłócenia.
Cyfrowy PCR trzeciej generacji
Cyfrowy PCR (DigitalPCR, dPCR, Dig-PCR) oblicza liczbę kopii sekwencji docelowej poprzez wykrywanie punktu końcowego i może przeprowadzać dokładne bezwzględne wykrywanie ilościowe bez stosowania kontroli wewnętrznych i krzywych wzorcowych.
Cyfrowy PCR wykorzystuje wykrywanie punktu końcowego i nie zależy od wartości Ct (progu cyklu), więc wydajność amplifikacji ma mniejszy wpływ na reakcję cyfrowego PCR, a tolerancja na inhibitory reakcji PCR jest lepsza, z dużą dokładnością i powtarzalnością.
Ze względu na charakterystykę wysokiej czułości i wysokiej dokładności nie jest łatwo zakłócany przez inhibitory reakcji PCR i może osiągnąć prawdziwą bezwzględną ocenę ilościową bez standardowych produktów, co stało się gorącym punktem badań i zastosowań.
Zgodnie z różnymi formami jednostki reakcyjnej można ją podzielić na trzy główne typy: układy mikroprzepływowe, wiórowe i kropelkowe.
1) Mikroprzepływowa cyfrowa PCR, mdPCR:
W oparciu o technologię mikroprzepływową rozdzielana jest matryca DNA.Technologia mikroprzepływowa może realizować nano-ulepszanie próbki lub generowanie mniejszych kropelek, ale kropelki wymagają specjalnej metody adsorpcji, a następnie połączenia z systemem reakcji PCR.mdPCR został stopniowo przyjęty przez inne metody.
2) Cyfrowy PCR na bazie kropli, ddPCR:
Użyj technologii generowania kropel wody w oleju, aby przetworzyć próbkę na kropelki i podzielić układ reakcyjny zawierający cząsteczki kwasu nukleinowego na tysiące kropelek w nanoskali, z których każda nie zawiera docelowej cząsteczki kwasu nukleinowego do wykrycia lub zawiera jedną do kilku docelowych cząsteczek kwasu nukleinowego do przetestowania.
3) Cyfrowy PCR oparty na chipie, cdPCR:
Wykorzystaj zintegrowaną technologię przepływu płynów do grawerowania wielu mikroprobówek i mikrownęk na płytkach krzemowych lub szkle kwarcowym i kontroluj przepływ roztworu przez różne zawory kontrolne, a także podziel próbkę cieczy na nanometry tego samego rozmiaru w dołkach reakcyjnych w celu przeprowadzenia cyfrowej reakcji PCR w celu uzyskania bezwzględnej oceny ilościowej.
Główne wady PCR trzeciej generacji:
Sprzęt i odczynniki są drogie.
Wymagania dotyczące jakości szablonu są wysokie.Jeśli liczba szablonów przekroczy liczbę mikrosystemów, nie będzie możliwe określenie ilościowe, a jeśli będzie zbyt mała, dokładność kwantyfikacji zostanie zmniejszona.
Fałszywie dodatnie wyniki mogą być również generowane w przypadku amplifikacji nieswoistej.
Czas postu: lipiec-30-2021