PCR, wiele PCR, PCR in situ, Odwrotny PCR, RT-PCR, qPCR(1)–PCR
Uporządkujemy koncepcje, kroki i szczegóły różnych PCR
Ⅰ. PCR
Reakcja łańcuchowa polimerazy, zwana PCR, to technologia biologii molekularnej, która służy do powiększania określonych fragmentów DNA.Można to uznać za szczególną replikację DNA in vitro.Polimerazę DNA (DNA Polymerase I) odkryto już w 1955 roku, a fragment Klenowa E. Coli, który ma wartość eksperymentalną i praktyczność, odkrył dr H. Klenow na początku lat 70.Stosowane obecnie enzymy (zwane polimerazą Taq) zostały wyizolowane z Thermus aquaticus, bakterii z gorących źródeł w 1976 roku. Charakteryzuje się odpornością na wysoką temperaturę i jest idealnym enzymem, ale jest szeroko stosowany po latach 80. XX wieku.Oryginalna koncepcja pierwotnego prymitywnego prototypu PCR jest podobna do naprawy i kopiowania genów, którą zaproponował dr KJell Kleppe w 1971 roku. Opublikował on pierwszą prostą i krótkoterminową kopię genu (podobną do reakcji dwóch pierwszych cykli PCR).Opracowany dzisiaj PCR został opracowany przez dr Kary'ego B. Mullisa w 1983 roku. Dr Mullis służył w tamtym roku firmom PE, więc PE ma specjalny status w branży PCR.Dr Mullis oficjalnie opublikował pierwszy powiązany artykuł wraz z Saiki i innymi w 1985 roku. Od tego czasu PCR stosuje się tysiące mil dziennie, a jakość powiązanych artykułów sprawia, że wiele innych metod badawczych jest niesmacznych.Następnie technologia PCR jest szeroko stosowana w biologicznych badaniach naukowych i zastosowaniach klinicznych, stając się najważniejszą technologią badań biologii molekularnej.Mullis zdobył także Nagrodę Nobla w dziedzinie chemii w 1993 roku.
PCRZasada
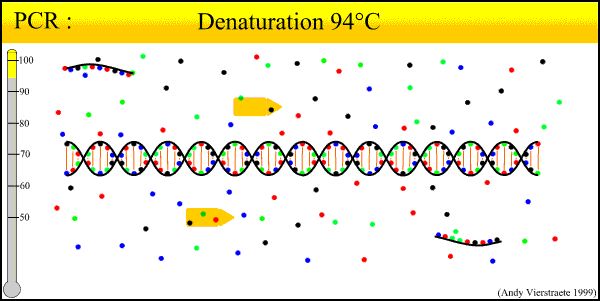
Podstawowa zasada technologii PCR jest podobna do naturalnego procesu replikacji DNA, a jej specyficzność zależy od startera oligonukleotydowego, który jest komplementarny do obu końców docelowej sekwencji.PCR składa się z trzech podstawowych etapów reakcji: degeneracja-wyżarzanie-wydłużenie: ①Degeneracja matrycy DNA: po podgrzaniu matrycy DNA do około 93°C przez określony czas, roztwór podwójnego DNA dla dwułańcuchowego DNA utworzonego przez amplifikację PCR matrycy DNA Opuszczanie tworzy pojedynczy łańcuch, dzięki czemu można go połączyć ze starterem w celu przygotowania do następnej rundy reakcji.② Wyżarzanie (związek) DNA matrycy i startera: Po podgrzaniu matrycy DNA i degeneracji w pojedynczy łańcuch temperatura spada do około 55°C.Komplementarna sekwencja startera i matrycy jednołańcuchowego DNA.③Przedłużenie startera: matryca DNA - wiązanie startera opiera się na działaniu polimerazy TaqDNA, z dNTP jako surowcem reakcji.Zachowaj zasadę replikacji, zsyntetyzuj nowy częściowo zarezerwowany łańcuch kopii, który uzupełnia szablonowy łańcuch DNA i powtórz cykl degeneracji-wyżarzania-przedłużenia trzy procesy mogą uzyskać więcej „częściowo zarezerwowanego łańcucha kopii”, a ten nowy łańcuch jest ponownie dostępny Zostań szablonem dla następnego cyklu.Zakończenie pętli zajmuje 2-4 minuty, docelowy gen może zostać zamplifikowany kilka milionów razy w ciągu 2-3 godzin.
StandardPCRSystem reakcji
| Polimeraza DNA Taq | 2,5 μl |
| Mg2+ | 1,5 mmol/L |
| 10x bufor do amplifikacji | 10 μl |
| 4 mieszaniny dNTP | 200 μl |
| Szablonowe DNA | 0,1 ~ 2 μg |
| Elementarz | 10 ~ 100 μl |
| Dodaj podwójną lub potrójną parującą wodę | 100 μl |
Pięć elementów reakcji PCR
W reakcji PCR bierze udział głównie pięć rodzajów substancji, a mianowicie starter, enzym, dNTP, matryca i bufor (wymagany jest Mg2+).[Procedura PCR]
Standardowy proces PCR dzieli się na trzy etapy
1. Degeneracja DNA (90°C-96°C): Dwułańcuchowe matryce DNA pod działaniem temperatury, wiązania wodorowe pękają, tworząc jednołańcuchowy DNA.
2. Wygrzewanie (25 ℃ -65 ℃): Temperatura systemu jest obniżana, starter jest łączony z matrycą DNA, tworząc lokalny podwójny łańcuch.
3. Wydłużenie (70 ℃ -75 ℃): Pod działaniem enzymu Taq (około 72 ° C, najlepsza aktywność), dNTP jest używany jako surowiec, rozciąga się od końca 5 'startera → koniec 3', synteza i szablon uzupełniają łańcuch DNA.
Każdy cykl jest denaturowany, wyżarzany i wydłużany, podwajając zawartość DNA.Obecnie, ze względu na krótki obszar amplifikacji, niektóre PCR mogą być replikowane w bardzo krótkim czasie, nawet jeśli aktywność enzymu Taq nie jest optymalna, więc można to zmienić na dwa etapy, to znaczy hybrydyzację i wydłużanie można przeprowadzić w 60°C-65°C w tym samym czasie.W celu ograniczenia procesu podnoszenia i chłodzenia oraz poprawy szybkości reakcji.
Funkcje reakcji PCR
● Wysoka specyficzność
Konkretnymi czynnikami decydującymi o odpowiedzi PCR są: ①Specyficzna kombinacja startera i matrycy DNA.② Zasada parowania zasad.③Lojalność reakcji syntezy polimerazy TaqDNA.④Specyficzność i konserwatywność docelowego genu.
Kluczem jest właściwa kombinacja starterów i szablonów.Wiązanie startera i matrycy oraz wydłużanie łańcucha startera opiera się na zasadzie dopasowania zasad alkalicznych.Lojalność reakcji syntezy polimerazy i odporność na wysoką temperaturę polimerazy Taq DNA do wiązania (związku) matrycy i startera w reakcji można przeprowadzić w wyższej temperaturze.Specyficzność kombinacji jest znacznie zwiększona.Klip może zachować wysoki stopień poprawności.Wybierając docelowy region genetyczny o wysokiej konserwatywności i wysokiej konserwatywności, jego specyficzność jest wyższa.
● Wysoka czułość
Wielkość produkcji produktów PCR zwiększa się o indeks, który może rozszerzyć szablon startowy Pickera (PG=10-12) do poziomu mikrokontrolera do poziomu mikrogramów (μg= -6).Komórki docelowe można wykryć z 1 miliona komórek;w wykrywaniu wirusów czułość PCR może osiągnąć 3 RFU (puste miejsca utworzone jednostki);minimalny wskaźnik wykrywalności w nauce o bakteriach to 3 bakterie.
● Prosty i szybki
Odbicie PCR wykorzystuje wysokotemperaturową polimerazę Taq DNA, która jednorazowo dodaje roztwór reakcyjny, czyli reakcję degeneracji-anneal-extension na roztwór do amplifikacji DNA i łaźnię wodną.Na ogół reakcja amplifikacji jest zakończona w ciągu 2 do 4 godzin.Produkty rozszerzone są generalnie analizowane za pomocą miecza elektrycznego i nie muszą używać izotopów, zanieczyszczeń radioaktywnych i łatwej promocji.
● Czystość próbki jest niska
Nie ma potrzeby oddzielania wirusów lub bakterii od komórek hodowlanych.Surowe produkty DNA i RNA mogą być używane jako wzmacniacze.Wykrywanie amplifikacji DNA można stosować bezpośrednio przy użyciu próbek klinicznych, takich jak krew, płyny ustrojowe, płyn do płukania kaszlu, włosy, komórki i żywe tkanki.
PCRczęste problemy
● Fałszywie negatywny, brak wzmocnionych prążków
Kluczowe etapy reakcji PCR to: ① przygotowanie matrycowych kwasów nukleinowych, ② jakość i specyficzność starterów, ③ jakość enzymów, ④ Warunki cyklu PCR.Znalezienie przyczyny należy również przeanalizować i przestudiować pod kątem powyższych linków.
Matryce: ① Matryca zawiera różne białka, ② Matryca zawiera inhibitor enzymu Taq, ③ Białko w matrycy nie jest eliminowane, zwłaszcza białko grupowe w chromosomie.⑤ Degeneracja kwasu nukleinowego Deminer nie jest dokładna.Gdy jakość enzymów i starterów jest dobra, nie ma prążka amplifikacji, co najprawdopodobniej jest spowodowane trawieniem próbek.Coś jest nie tak z matrycowym procesem ekstrakcji kwasu nukleinowego, więc aby przygotować skuteczny i stabilny roztwór do trawienia, jego procedura powinna być ustalona, a nie dowolnie zmieniana.
Inaktywacja enzymu: nowy enzym lub stare i nowe enzymy należy stosować razem, aby przeanalizować, czy aktywność enzymu jest utracona lub niewystarczająca, co prowadzi do wyników fałszywie ujemnych.Należy zauważyć, że czasami zapomina się o enzymie Taq lub bromku etydyny.
Primer: jakość startera, stężenie startera i czy stężenie dwóch starterów jest symetryczne.Jest to częsta przyczyna niepowodzenia reakcji PCR lub rosnący prążek nie jest idealny i ma skłonność do dyfuzji.Występują problemy z jakością podkładów niektórych numerów partii.Oba startery mają wysokie i niskie stężenie, powodując asymetryczną amplifikację o niskiej wydajności.Środki zaradcze to: ① Wybierz dobry starter do syntezy jednostek.② Stężenie startera nie tylko zależy od wartości OD, ale także zwraca uwagę na oryginalny płyn startera do elektroforezy w żelu z cukru agarowego.Musi istnieć strefa paska podkładu, a jasność dwóch podkładów powinna być ogólnie spójna.Belt, PCR może w tym momencie zawieść i należy go rozwiązać za pomocą jednostki syntezy starterów.Jeśli podkład jest wysoki, jasność jest niska, a jego stężenie musi być zrównoważone po rozcieńczeniu.③ Primer powinien być opłacony i przechowywany w wysokim stężeniu, aby zapobiec wielokrotnemu zamarzaniu lub długotrwałemu chłodzeniu części lodówki, co spowoduje zniszczenie i degradację primera.④ Projekt startera jest nierozsądny, na przykład długość startera jest niewystarczająca, a klaster di tworzy się między starterami.
Stężenie Mg2+: Stężenie jonów Mg2+ ma duży wpływ na wydajność amplifikacji PCR.Nadmierne stężenie może zmniejszyć płeć przeciwną amplifikacji PCR.Jeśli stężenie jest zbyt niskie, wyjście amplifikacji PCR spowoduje nawet niepowodzenie amplifikacji PCR bez prążka ekspansji.
Zmiana objętości reakcji: Objętość stosowana w amplifikacji PCR wynosi 20 ul, 30 ul i 50 ul lub 100 ul, duża objętość aplikacji do amplifikacji PCR jest ustalana zgodnie z różnymi celami badań naukowych i testów klinicznych.Po wykonaniu małych objętości, takich jak 20 ul, konieczne jest wykonanie warunku sznurka podczas tworzenia rozmiaru, w przeciwnym razie zawiedzie.
Przyczyny fizyczne: Transformacja jest bardzo ważna dla amplifikacji PCR.Jeśli temperatura degeneracji jest niska, czas degeneracji jest krótki, prawdopodobnie wystąpi w przypadku wyników fałszywie ujemnych;zbyt niska temperatura wygrzewania może powodować niespecyficzną amplifikację i zmniejszać specyficzną skuteczność amplifikacji.Silnie wpływają na kombinację starterów i matryc, aby zmniejszyć wydajność amplifikacji PCR.Czasami konieczne jest użycie standardowych termometrów do wykrycia zmienności, wyżarzania i rozszerzonej temperatury w przedłużaczu lub warniku rozpuszczalnym w wodzie, co jest jedną z przyczyn niepowodzenia reakcji PCR.
Warianty sekwencji docelowej: jeśli występuje sekwencja docelowa, mutacja lub delecja, połączenie prototypu i matrycy jest łączone lub z powodu braku sekwencji docelowej starter i matryca utracą sekwencję komplementarną, a amplifikacja PCR nie zakończy się sukcesem.
● Fałszywie pozytywny
Prążek amplifikacji PCR wydaje się zgodny z prążkiem sekwencji docelowej, a czasami jego prążek jest bardziej wyrównany i wyższy.
Projekt startera nie jest odpowiedni: wybrana sekwencja amplifikacji i niecelowa sekwencja amplifikacji są homologiczne, więc podczas amplifikacji PCR produkty amplifikacji PCR są sekwencjami niecelowymi.Sekwencja docelowa jest zbyt krótka lub starter jest zbyt krótki i jest podatny na fałszywie dodatni wynik.Trzeba przeprojektować.
Zanieczyszczenie krzyżowe docelowej sekwencji lub produktów amplifikacji: Istnieją dwa powody tego zanieczyszczenia: Po pierwsze, zanieczyszczenie krzyżowe całego genomu lub dużych segmentów, co prowadzi do fałszywych alarmów.Ten rodzaj fałszywie dodatnich wyników można rozwiązać następującymi metodami: Zachowaj ostrożność i delikatność podczas pracy, aby zapobiec wdychaniu docelowej sekwencji do pistoletu do pobierania próbek lub rozpryskiwaniu się probówki wirówkowej.Z wyjątkiem enzymów i substancji, które nie wytrzymują wysokich temperatur, wszystkie odczynniki lub sprzęt należy dezynfekować pod wysokim ciśnieniem.Rury wirówkowe i próbki powinny być używane jednorazowo.W razie potrzeby, przed dodaniem próbek, probówkę reakcyjną i odczynnik wystawia się na działanie promieni ultrafioletowych w celu zniszczenia istniejącego kwasu nukleinowego.Po drugie, małe fragmenty w zanieczyszczeniu powietrza.Te małe fragmenty są krótsze niż sekwencja docelowa, ale wykazują pewną homologię.Można go łączyć ze sobą.Po uzupełnieniu starterów produkt PCR może ulec ekspansji, co spowoduje fałszywie dodatnią produkcję.Może być wykorzystany do zredukowania lub wyeliminowania metody nest PCR.
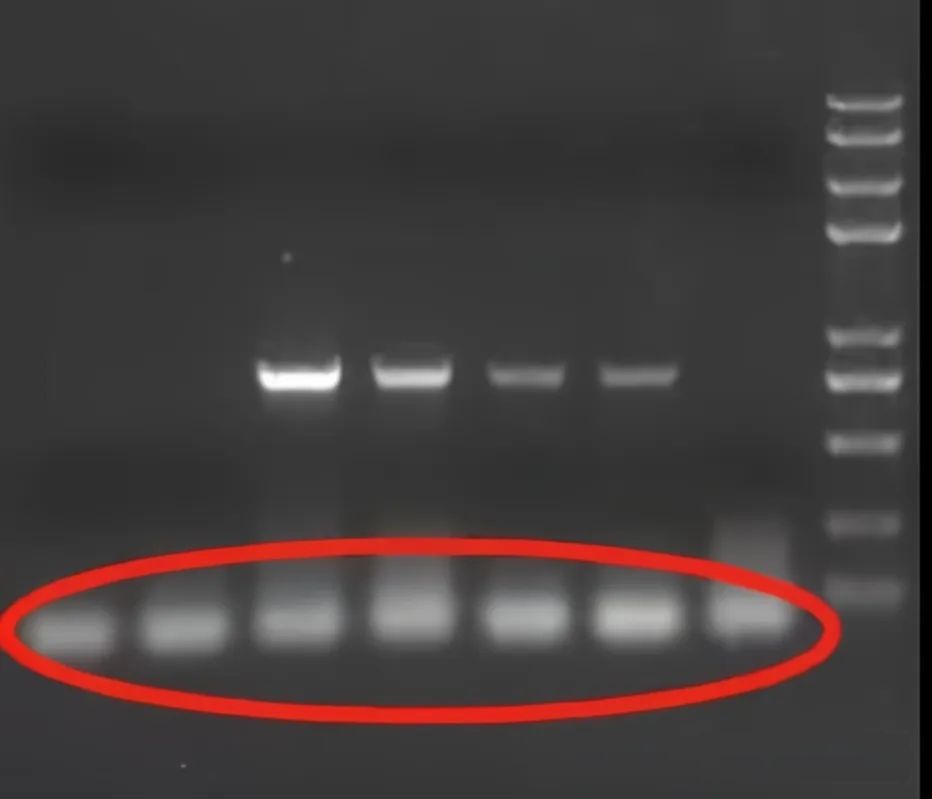
● Pojawiają się niespecyficzne prążki amplifikacji
Prążki, które pojawiły się po amplifikacji PCR, są niespójne z oczekiwaną wielkością, albo duże, albo małe, albo jednocześnie lub w tym samym czasie, swoiste prążki amplifikacji i nieswoiste prążki amplifikacji.Pojawienie się niespecyficznych prążków jest następujące: Po pierwsze, startery są niekompletnie komplementarne do sekwencji docelowej lub polimeryzacja startera z utworzeniem klastra di.Po drugie, stężenie jonów MG2+ jest zbyt wysokie, temperatura hybrydyzacji jest zbyt niska, a liczba cykli PCR jest powiązana.Po drugie, jakość i ilość enzymów.Często enzymy z niektórych źródeł są podatne na niespecjalne prążki, a enzymy z innego źródła nie występują.Czasami dochodzi również do niespecyficznej amplifikacji enzymów.Środki zaradcze to: przeprojektowanie atrakcji, jeśli to konieczne.Zmniejsz ilość enzymu lub wymień enzym z innego źródła.Zmniejszyć ilość pierwotnego, odpowiednio zwiększyć liczbę szablonów i zmniejszyć liczbę cykli.Odpowiednio zwiększyć temperaturę wyżarzania lub zastosować metodę dwóch punktów temperaturowych (degeneracja 93°C, wyżarzanie i wydłużanie w temperaturze około 65°C).
● Pojawiają się łuszczące się pakuły lub taśma rozmazująca
Czasami wydaje się, że amplifikacja PCR jest nakładana, łuskana lub przypominająca dywan.Z tego powodu, z powodu nadmiernej ilości enzymów lub złej jakości enzymu, stężenie dNTP jest zbyt wysokie, stężenie Mg2+ jest zbyt wysokie, temperatura wygrzewania jest zbyt niska, a liczba cykli jest zbyt duża.Środki zaradcze to: ①Zmniejsz ilość enzymów lub zmień enzym z innego źródła.②Zmniejsz stężenie dNTP ③Odpowiednio zredukuj stężenie Mg2+.④Zwiększ liczbę szablonów i zmniejsz liczbę cykli.
Produkty powiązane
◮ Większa wierność: 6 razy większa niż w przypadku zwykłego enzymu Taq;
◮ Większa prędkość amplifikacji
◮ Większe możliwości adaptacji szablonów
◮ Wyższa wydajność wzmocnienia
◮ Większa tolerancja środowiskowa: umieszczone w temperaturze 37°C przez tydzień, zachowując ponad 90% aktywności;
◮ Ma aktywność polimerazy DNA 5'→3' i aktywność egzonukleazy 5'→3', bez aktywności egzonukleazy 3'→5'.
Unikalny system reakcji oraz wysokowydajna polimeraza DNA Taq sprawiają, że reakcja PCR charakteryzuje się wyższą wydajnością amplifikacji, specyficznością i czułością.
RT-qPCR Easyᵀᴹ (One Step)-SYBR Green I
◮ Jednoetapowy zestaw umożliwia przeprowadzenie odwrotnej transkrypcji i qPCR w dwóch reakcjach w tej samej probówce, wystarczy dodać matrycę RNA, specyficzne startery do PCR i wolne od RNazy ddH2O.
◮ Zestaw umożliwia szybką i wydajną analizę ilościową wirusowego RNA lub śladowego RNA.
◮ Zestaw wykorzystuje unikalny odczynnik Foregene do odwrotnej transkrypcji i polimerazę Foregene HotStar Taq DNA w połączeniu z unikalnym systemem reakcji, aby skutecznie poprawić wydajność amplifikacji i specyficzność reakcji.
◮ Zoptymalizowany system reakcji sprawia, że reakcja ma wyższą czułość wykrywania, większą stabilność termiczną i lepszą tolerancję.
◮ Łatwy RT-qPCRTM(One Step)-SYBR Green I kit jest dostarczany z wewnętrznym barwnikiem referencyjnym ROX, którego można użyć do wyeliminowania tła sygnału i błędów sygnału między dołkami, co jest wygodne dla klientów w użyciu w różnych modelach ilościowych urządzeń do PCR.
RT ŁatweTMII (Mistrzowy Premiks dla synteza pierwszej nici cDNA dlaPCR w czasie rzeczywistym)
-Wydajna zdolność do usuwania gDNA, która może usunąć gDNA z szablonu w ciągu 2 minut.
-Wydajny system odwrotnej transkrypcji, synteza pierwszej nici cDNA zajmuje tylko 15 minut.
-Złożone szablony: szablony o wysokiej zawartości GC i złożonej strukturze drugorzędowej można również odwracać z dużą wydajnością.
- System odwrotnej transkrypcji o wysokiej czułości, szablony na poziomie pg mogą również uzyskać wysokiej jakości cDNA.
- System odwrotnej transkrypcji ma wysoką stabilność termiczną, optymalna temperatura reakcji wynosi 42 ℃ i nadal ma dobrą wydajność odwrotnej transkrypcji przy 50 ℃.
Czas postu: 18 marca 2023 r