- PCR to metoda stosowana do amplifikacji DNA z małej ilości matrycy DNA.RT-PCR wykorzystuje odwrotną transkrypcję w celu wytworzenia matrycy DNA ze źródła RNA, którą można następnie amplifikować.
- PCR i RT-PCR są zazwyczaj reakcjami punktu końcowego, podczas gdy qPCR i RT-qPCR wykorzystują kinetykę szybkości syntezy produktu podczas reakcji PCR do ilościowego określenia ilości obecnej matrycy.
- Nowsze metody, takie jak cyfrowa PCR, umożliwiają bezwzględne oznaczenie ilościowe początkowej matrycy DNA, podczas gdy metody takie jak izotermiczna PCR zmniejszają potrzebę stosowania drogiego sprzętu w celu zapewnienia wiarygodnych wyników.
Reakcja łańcuchowa polimerazy (PCR) to stosunkowo prosta i szeroko stosowana technika biologii molekularnej służąca do amplifikacji i wykrywania sekwencji DNA i RNA.W porównaniu z tradycyjnymi metodami klonowania i amplifikacji DNA, które często mogą trwać kilka dni, PCR wymaga tylko kilku godzin.PCR jest bardzo czuły i wymaga minimalnej matrycy do wykrywania i amplifikacji określonych sekwencji.Podstawowe metody PCR rozwinęły się dalej od prostego wykrywania DNA i RNA.Poniżej przedstawiamy przegląd różnych metod PCR i odczynników, które dostarczamy w Enzo Life Sciences na potrzeby badań.Naszym celem jest pomóc naukowcom w szybkim dostępie do odczynników PCR do wykorzystania w kolejnym projekcie badawczym!
PCR
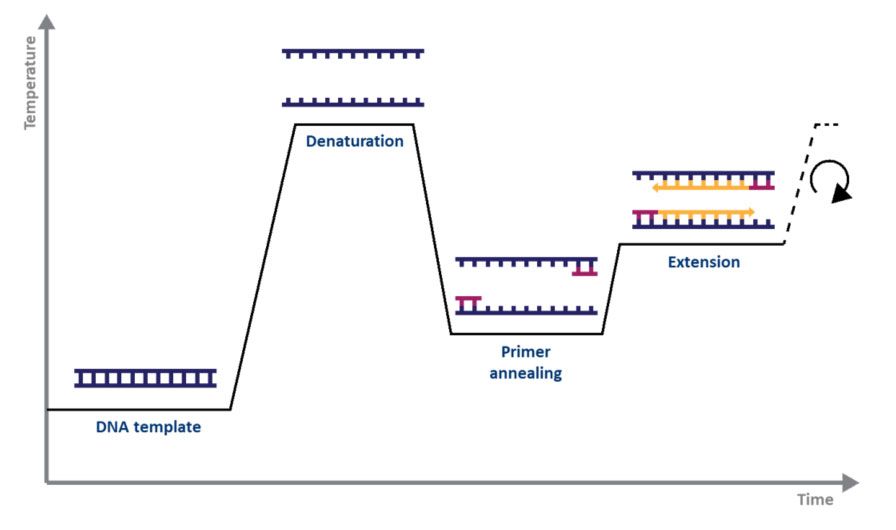
Do standardowej PCR wystarczy polimeraza DNA, magnez, nukleotydy, startery, matryca DNA do amplifikacji i termocykler.Mechanizm PCR jest tak prosty, jak jego cel: 1) dwuniciowy DNA (dsDNA) ulega denaturacji cieplnej, 2) startery dopasowują się do pojedynczych nici DNA oraz 3) startery są wydłużane przez polimerazę DNA, w wyniku czego powstają dwie kopie pierwotna nić DNA.Proces denaturacji, wyżarzania i wydłużania w szeregu temperatur i czasów nazywany jest jednym cyklem amplifikacji (ryc. 1).
| Rysunek 1.Schematyczne przedstawienie cyklu amplifikacji metodą PCR. |
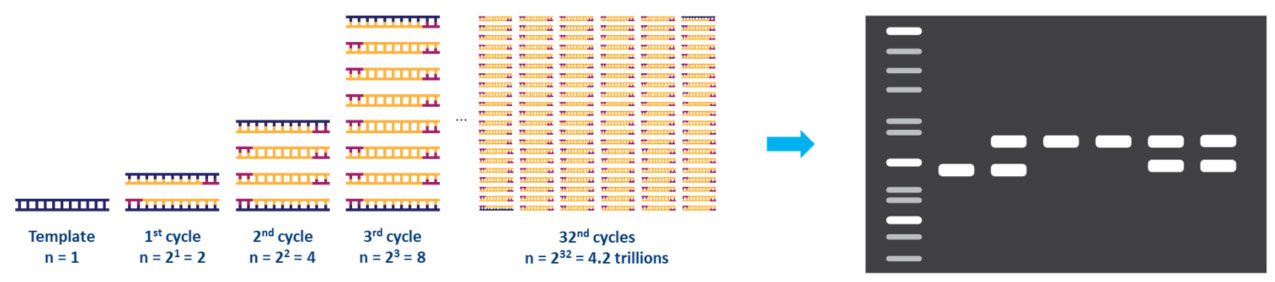
Każdy etap cyklu powinien być zoptymalizowany pod kątem użytego zestawu szablonów i podkładów.Cykl ten powtarza się około 20–40 razy, a amplifikowany produkt można następnie poddać analizie, zazwyczaj w żelu agarozowym (ryc. 2).
| Rysunek 2.Amplifikacja matrycy DNA metodą PCR i analiza metodą elektroforezy w żelu agarozowym. |
Ponieważ PCR jest metodą bardzo czułą i do pojedynczych reakcji potrzebne są bardzo małe objętości, zaleca się przygotowanie master mixu na kilka reakcji.Master mix musi być dobrze wymieszany, a następnie podzielony przez liczbę reakcji, upewniając się, że każda reakcja będzie zawierać tę samą ilość enzymu, dNTP i starterów.Wielu dostawców, takich jak Enzo Life Sciences, oferuje również mieszanki PCR, które zawierają już wszystko oprócz starterów i matrycy DNA.
Regiony bogate w guaninę/cytozynę (bogate w GC) stanowią wyzwanie w standardowych technikach PCR.Sekwencje bogate w GC są bardziej stabilne niż sekwencje z niższą zawartością GC.Ponadto sekwencje bogate w GC mają tendencję do tworzenia struktur drugorzędowych, takich jak pętle typu spinka do włosów.W rezultacie podwójne nici bogate w GC są trudne do całkowitego oddzielenia w fazie denaturacji.W konsekwencji polimeraza DNA nie może syntetyzować nowej nici bez przeszkód.Wyższa temperatura denaturacji może to poprawić, a dostosowanie w kierunku wyższej temperatury przyłączania i krótszego czasu przyłączania może zapobiec niespecyficznemu wiązaniu starterów bogatych w GC.Dodatkowe odczynniki mogą zwiększyć amplifikację sekwencji bogatych w GC.DMSO, glicerol i betaina pomagają rozbić struktury drugorzędowe powstałe w wyniku interakcji GC, ułatwiając w ten sposób rozdzielenie podwójnych nici.
PCR na gorąco
Niespecyficzna amplifikacja jest problemem, który może wystąpić podczas PCR.Większość polimeraz DNA stosowanych w PCR działa najlepiej w temperaturach od 68°C do 72°C.Enzym może jednak działać także w niższych temperaturach, chociaż w mniejszym stopniu.W temperaturach znacznie niższych od temperatury przyłączania startery mogą wiązać się niespecyficznie i prowadzić do niespecyficznej amplifikacji, nawet jeśli reakcja przebiega na lodzie.Można temu zapobiec, stosując inhibitory polimerazy, które dysocjują od polimerazy DNA dopiero po osiągnięciu określonej temperatury, stąd termin PCR z gorącym startem.Inhibitorem może być przeciwciało, które wiąże polimerazę i powoduje denaturację w początkowej temperaturze denaturacji (typowo 95°C).
Polimeraza o wysokiej wierności
Chociaż polimerazy DNA amplifikują się dość dokładnie do pierwotnej sekwencji matrycy, mogą wystąpić błędy w dopasowaniu nukleotydów.Niedopasowania w zastosowaniach takich jak klonowanie mogą skutkować skróconymi transkryptami i błędnie przetłumaczonymi lub nieaktywnymi białkami w dalszej części.Aby uniknąć tych rozbieżności, zidentyfikowano i włączono do procesu polimerazy o działaniu „korekty”.Pierwszą polimerazę korygującą, Pfu, zidentyfikowano w 1991 roku u Pyrococcus furiosus.Ten enzym Pfu ma aktywność egzonukleazy 3' do 5'.W miarę amplifikacji DNA egzonukleaza usuwa niedopasowane nukleotydy na końcu 3' nici.Następnie zostaje zastąpiony prawidłowy nukleotyd i synteza DNA jest kontynuowana.Identyfikacja nieprawidłowych sekwencji nukleotydowych opiera się na powinowactwie wiązania prawidłowego trifosforanu nukleozydu z enzymem, gdzie nieefektywne wiązanie spowalnia syntezę i pozwala na prawidłową wymianę.Aktywność korygująca polimerazy Pfu powoduje mniej błędów w końcowej sekwencji w porównaniu z polimerazą DNA Taq.W ostatnich latach zidentyfikowano inne enzymy korygujące i wprowadzono modyfikacje oryginalnego enzymu Pfu w celu dalszego zmniejszenia poziomu błędów podczas amplifikacji DNA.
RT-PCR
PCR z odwrotną transkrypcją, czyli RT-PCR, pozwala na użycie RNA jako matrycy.Dodatkowy etap pozwala na detekcję i amplifikację RNA.RNA ulega odwrotnej transkrypcji do komplementarnego DNA (cDNA) przy użyciu odwrotnej transkryptazy.Jakość i czystość matrycy RNA są niezbędne dla powodzenia RT-PCR.Pierwszym etapem RT-PCR jest synteza hybrydy DNA/RNA.Odwrotna transkryptaza ma również funkcję RNazy H, która degraduje część RNA hybrydy.Jednoniciowa cząsteczka DNA jest następnie uzupełniana przez zależną od DNA aktywność polimerazy DNA poprzez odwrotną transkryptazę do cDNA.Wydajność reakcji pierwszej nici może mieć wpływ na proces amplifikacji.Od tego momentu do amplifikacji cDNA stosowana jest standardowa procedura PCR.Możliwość przekształcenia RNA w cDNA metodą RT-PCR ma wiele zalet i jest wykorzystywana przede wszystkim do analizy ekspresji genów.RNA jest jednoniciowy i bardzo niestabilny, co utrudnia pracę z nim.Zwykle służy jako pierwszy etap w qPCR, która określa ilościowo transkrypty RNA w próbce biologicznej.
qPCR i RT-qPCR
Ilościowa PCR (qPCR) służy do wykrywania, charakteryzowania i oznaczania ilościowego kwasów nukleinowych w wielu zastosowaniach.W metodzie RT-qPCR transkrypty RNA często oznacza się ilościowo, najpierw dokonując odwrotnej transkrypcji na cDNA, jak opisano powyżej, a następnie przeprowadza się qPCR.Podobnie jak w standardowej PCR, DNA jest amplifikowane w trzech powtarzających się etapach: denaturacja, hybrydyzacja i elongacja.Jednakże w qPCR znakowanie fluorescencyjne umożliwia gromadzenie danych w miarę postępu PCR.Technika ta ma wiele zalet ze względu na zakres dostępnych metod i środków chemicznych.
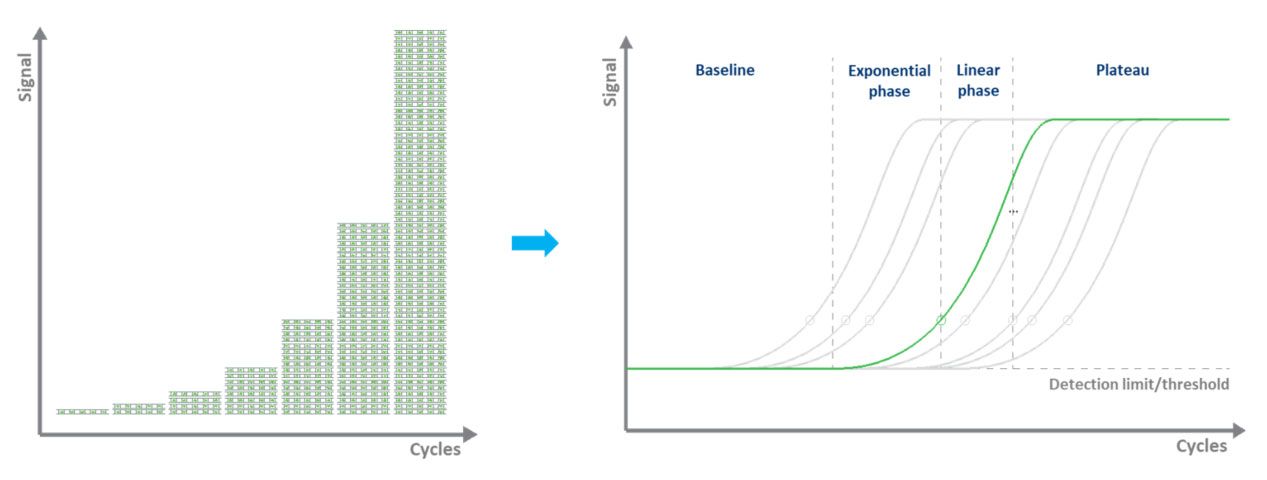
W qPCR opartej na barwniku (zazwyczaj zielonym), znakowanie fluorescencyjne umożliwia ilościowe oznaczenie amplifikowanych cząsteczek DNA poprzez zastosowanie barwnika wiążącego dsDNA.Podczas każdego cyklu mierzona jest fluorescencja.Sygnał fluorescencji wzrasta proporcjonalnie do ilości replikowanego DNA.Dlatego też ilość DNA jest oznaczana „w czasie rzeczywistym” (ryc. 3).Wadą qPCR opartej na barwniku jest to, że jednocześnie można badać tylko jeden cel i że barwnik wiąże się z dowolnym ds-DNA obecnym w próbce.
| Rysunek 3.Amplifikacja matrycy DNA metodą qPCR i pomiar sygnału fluorescencji w czasie rzeczywistym. |
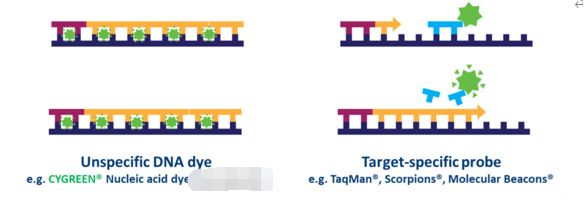
W metodzie qPCR opartej na sondzie można wykryć wiele obiektów docelowych jednocześnie w każdej próbce, ale wymaga to optymalizacji i zaprojektowania sond(y) specyficznych dla celu, stosowanych oprócz starterów.Dostępnych jest kilka typów sond, ale najpowszechniejszym typem jest sonda do hydrolizy, która zawiera fluorofor i wygaszacz.Transfer energii rezonansu fluorescencji (FRET) zapobiega emisji fluoroforu przez wygaszacz, gdy sonda jest nienaruszona.Jednakże podczas reakcji PCR sonda ulega hydrolizie podczas wydłużania startera i amplifikacji specyficznej sekwencji, z którą jest związana.Rozszczepienie sondy oddziela fluorofor od środka wygaszającego i skutkuje zależnym od amplifikacji wzrostem fluorescencji (ryc. 4).Zatem sygnał fluorescencji z reakcji qPCR z użyciem sondy jest proporcjonalny do ilości docelowej sekwencji sondy obecnej w próbce.Ponieważ qPCR oparta na sondzie jest bardziej specyficzna niż qPCR oparta na barwniku, często jest to technologia stosowana w testach diagnostycznych opartych na qPCR.
| Rysunek 4.Różnice między qPCR opartym na barwniku i sondzie. |
Wzmocnienie izotermiczne
Wspomniane powyżej techniki PCR wymagają drogiego sprzętu do termocyklingu, aby dokładnie zwiększać i obniżać temperaturę w komorze na etapach denaturacji, przyłączania i wydłużania.Opracowano szereg technik, które nie wymagają tak precyzyjnych urządzeń i można je przeprowadzić w zwykłej łaźni wodnej lub nawet w odpowiednich komórkach.Techniki te są wspólnie nazywane amplifikacją izotermiczną i działają w oparciu o wzmocnienie wykładnicze, liniowe lub kaskadowe.
Najbardziej znanym typem amplifikacji izotermicznej jest amplifikacja izotermiczna za pośrednictwem pętli, w skrócie LAMP.LAMP wykorzystuje amplifikację wykładniczą w temperaturze 65⁰C w celu amplifikacji matrycowego DNA lub RNA.Podczas wykonywania LAMP stosuje się cztery do sześciu starterów komplementarnych do regionów docelowego DNA z polimerazą DNA w celu syntezy nowego DNA.Dwa z tych starterów mają komplementarne sekwencje, które rozpoznają sekwencje w innych starterach i wiążą je, umożliwiając utworzenie struktury „pętli” w nowo zsyntetyzowanym DNA, która następnie wspomaga hybrydyzację starterów w kolejnych rundach amplifikacji.LAMPę można wizualizować wieloma metodami, w tym fluorescencją, elektroforezą w żelu agarozowym lub kolorymetrią.Łatwość wizualizacji i wykrywania obecności lub braku produktu za pomocą kolorymetrii oraz brak wymaganego drogiego sprzętu sprawiły, że LAMP stała się odpowiednią opcją do testowania SARS-CoV-2 w obszarach, gdzie kliniczne testy laboratoryjne nie były łatwo dostępne lub do przechowywania i transportu próbek nie było wykonalne lub w laboratoriach, które wcześniej nie posiadały sprzętu do termocyklingu PCR.
Czas publikacji: 19 sierpnia 2023 r