Materiał wyjściowy: RNA
Ilościowy PCR z odwrotną transkrypcją (RT-qPCR) to metoda eksperymentalna stosowana w eksperymentach PCR z wykorzystaniem RNA jako materiału wyjściowego.W tej metodzie całkowity RNA lub informacyjny RNA (mRNA) jest najpierw transkrybowany do komplementarnego DNA (cDNA) przez odwrotną transkryptazę.Następnie przeprowadzono reakcję qPCR z użyciem cDNA jako matrycy.RT-qPCR był używany w różnych zastosowaniach biologii molekularnej, w tym w analizie ekspresji genów, walidacji interferencji RNA, walidacji mikromacierzy, wykrywaniu patogenów, testach genetycznych i badaniach nad chorobami.
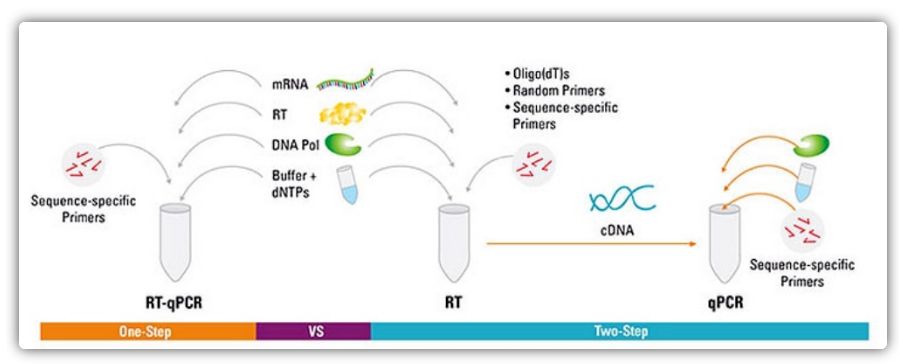
Jednoetapowe i dwuetapowe metody RT-qPCR
RT-qPCR można przeprowadzić metodą jednoetapową lub dwuetapową.Jednoetapowy RT-qPCR łączy odwrotną transkrypcję i amplifikację PCR, umożliwiając zakończenie reakcji odwrotnej transkryptazy i polimerazy DNA w tej samej probówce w tych samych warunkach buforowych.Jednoetapowy RT-qPCR wymaga jedynie użycia starterów specyficznych dla sekwencji.W dwuetapowym RT-qPCR odwrotną transkrypcję i amplifikację PCR przeprowadza się w dwóch probówkach, stosując różne zoptymalizowane bufory, warunki reakcji i strategie projektowania starterów.
| Korzyść | Niekorzyść | |
| Jeden krok | Ta metoda ma mniej błędów eksperymentalnych, ponieważ obie reakcje przeprowadza się w jednej probówce
Mniej etapów pipetowania zmniejsza ryzyko zanieczyszczenia
Nadaje się do amplifikacji/prześwietlania o dużej przepustowości, szybki i powtarzalny | Reakcji dwuetapowych nie można optymalizować oddzielnie
Ponieważ warunki reakcji są zagrożone przez połączenie reakcji dwuetapowej, czułość nie jest tak dobra jak w przypadku metody dwuetapowej
Liczba celów wykrytych przez pojedynczą próbkę jest niewielka |
| Dwa kroki | Możliwość tworzenia stabilnych bibliotek cDNA, które można przechowywać przez długi czas i wykorzystywać w wielu reakcjach
Geny docelowe i geny referencyjne można amplifikować z tej samej biblioteki cDNA bez potrzeby stosowania wielu bibliotek cDNA
Bufory reakcyjne i warunki reakcji umożliwiające optymalizację pojedynczych przebiegów reakcji
Elastyczny wybór warunków wyzwalania | Używanie wielu probówek i więcej etapów pipetowania zwiększa ryzyko zanieczyszczenia DNA, i czasochłonne.
Wymaga większej optymalizacji niż metoda jednoetapowa |
Produkty powiązane:
RT-qPCR Easyᵀᴹ (jeden krok)-SYBR Green I
RT-qPCR Easyᵀᴹ (One Step)-Taqman
RT Easyᴹ I Master Premix do syntezy pierwszej nici CDNA
Zestaw Real Time PCR Easyᵀᴹ-SYBR Green I
Selekcja całkowitego RNA i mRNA
Podczas projektowania eksperymentu RT-qPCR ważne jest, aby zdecydować, czy użyć całkowitego RNA, czy oczyszczonego mRNA jako matrycy do odwrotnej transkrypcji.Chociaż mRNA może być w stanie zapewnić nieco wyższą czułość, nadal często stosuje się całkowity RNA.Powodem tego jest to, że całkowity RNA ma ważniejszą przewagę jako materiał wyjściowy niż mRNA.Po pierwsze, proces wymaga mniejszej liczby etapów oczyszczania, co zapewnia lepsze ilościowe odzyskiwanie matrycy i lepszą normalizację wyników do początkowej liczby komórek.Po drugie, unika etapu wzbogacania mRNA, co pozwala uniknąć możliwości wypaczenia wyników z powodu różnych odzysków różnych mRNA.Ogólnie rzecz biorąc, ponieważ w większości zastosowań względna ocena ilościowa docelowego genu jest ważniejsza niż bezwzględna czułość wykrywania, w większości przypadków bardziej odpowiedni jest całkowity RNA.
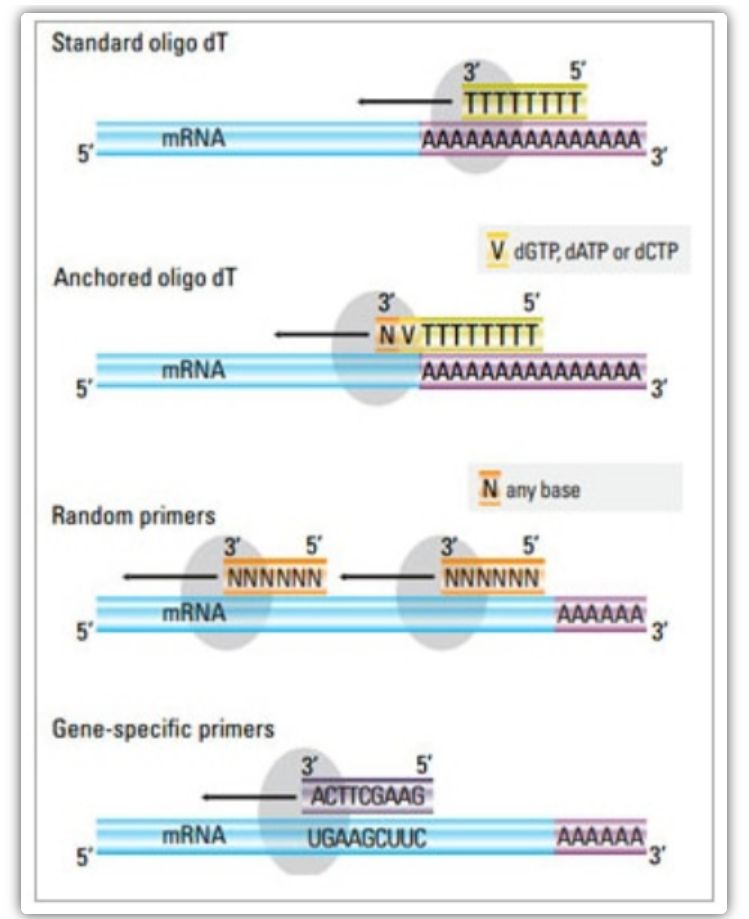
Starter odwrotnej transkrypcji
W metodzie dwuetapowej trzy różne metody mogą być stosowane do primowania reakcji cDNA: startery oligo(dT), startery losowe lub startery specyficzne dla sekwencji.Zazwyczaj stosuje się kombinację starterów oligo(dT) i starterów losowych.Te startery przyłączają się do matrycowej nici mRNA i dostarczają odwrotnej transkryptazy z punktem wyjścia do syntezy.
| Wybór podkładu | Struktura i funkcja | Korzyść | Niekorzyść |
| Starter oligo(dT) (lub zakotwiczony starter oligo(dT)) | Wydłużone przyłączanie do reszt tyminy na ogonie poli(A) mRNA;starter kotwicy oligo(dT) zawiera G, C lub A na końcu 3 '(miejsce zakotwiczenia) | Synteza pełnej długości cDNA z poli(A)-ogoniastego mRNA
Ma zastosowanie, gdy dostępna jest mniejsza ilość materiału wyjściowego
Miejsce zakotwiczenia zapewnia, że starter oligo(dT) wiąże się z ogonem poli(A) 5' mRNA | Nadaje się tylko do amplifikacji genów z ogonami poli(A).
Uzyskaj skrócony cDNA z miejsca pierwotnego*2 w poli(A)
Skierowany do wiązania się z końcem 3 '*
*Ta możliwość jest zminimalizowana, jeśli stosuje się zakotwiczone startery oligo(dT). |
| losowy podkład
| Długość od 6 do 9 zasad, które mogą przyłączać się do wielu miejsc podczas transkrypcji RNA | Anneal do wszystkich RNA (tRNA, rRNA i mRNA)
Nadaje się do transkryptów o znaczącej strukturze drugorzędowej lub gdy dostępny jest mniej materiału wyjściowego
Wysoka wydajność cDNA | cDNA jest poddawany odwrotnej transkrypcji z całego RNA, co zwykle nie jest pożądane i może osłabić sygnał docelowego mRNA
uzyskać skrócone cDNA |
| startery specyficzne dla sekwencji | Niestandardowe startery celujące w określone sekwencje mRNA | specyficzna biblioteka cDNA
Popraw czułość
Używanie starterów do odwrotnej reakcji qPCR | Ogranicza się tylko do syntezy pojedynczego genu docelowego |
Odwrotna transkryptaza
Odwrotna transkryptaza to enzym, który wykorzystuje RNA do syntezy DNA.Niektóre odwrotne transkryptazy mają aktywność RNazy i mogą degradować nici RNA w hybrydowych niciach RNA-DNA po transkrypcji.Jeśli nie ma aktywności enzymatycznej RNazy, można dodać RNazę H w celu uzyskania wyższej wydajności qPCR.Powszechnie stosowane enzymy obejmują odwrotną transkryptazę wirusa mysiej białaczki Moloneya i odwrotną transkryptazę wirusa ptasiego szpiczaka.W przypadku RT-qPCR idealnie jest wybrać odwrotną transkryptazę o wyższej termostabilności, aby synteza cDNA mogła być prowadzona w wyższych temperaturach, zapewniając pomyślną transkrypcję RNA o wyższej strukturze drugorzędowej, przy jednoczesnym zachowaniu ich pełnej aktywności przez cały czas reakcji, co skutkuje wyższymi wydajnościami cDNA.
Produkty powiązane:
Odwrotna transkryptaza Foreasy M-MLV
Aktywność RNazy H odwrotnej transkryptazy
RNazaH jest zdolna do degradacji nici RNA z dupleksów RNA-DNA, umożliwiając wydajną syntezę dwuniciowego DNA.Jednak przy użyciu długiego mRNA jako matrycy, RNA może ulec przedwczesnej degradacji, co skutkuje skróceniem cDNA.Dlatego często korzystne jest zminimalizowanie aktywności RNazyH podczas klonowania cDNA, jeśli pożądana jest synteza długich transkryptów.Natomiast odwrotne transkryptazy o aktywności RNazy H są często korzystne w zastosowaniach qPCR, ponieważ zwiększają topnienie dupleksów RNA-DNA podczas pierwszego cyklu PCR.
Projekt podkładu
Startery do PCR stosowane na etapie qPCR w RT-qPCR powinny być idealnie zaprojektowane tak, aby obejmowały połączenie egzon-ekson, gdzie starter do amplifikacji mógłby potencjalnie obejmować rzeczywistą granicę ekson-intron.Ponieważ sekwencje genomowego DNA zawierające introny nie są amplifikowane, ten projekt zmniejsza ryzyko fałszywie dodatnich wyników amplifikacji z zanieczyszczenia genomowego DNA.
Jeśli nie można zaprojektować starterów w celu oddzielenia eksonów lub granic egzon-ekson, może być konieczne potraktowanie próbek RNA wolną od RNazy DNazą I lub dsDNazą w celu usunięcia zanieczyszczenia genomowym DNA.
Kontrola RT-qPCR
Kontrola negatywna odwrotnej transkrypcji (kontrola -RT) powinna być uwzględniona we wszystkich eksperymentach RT-qPCR w celu wykrycia zanieczyszczenia DNA (takiego jak genomowy DNA lub produkty PCR z poprzednich reakcji).Ta kontrola zawiera wszystkie składniki reakcji z wyjątkiem odwrotnej transkryptazy.Ponieważ w przypadku tej kontroli nie zachodzi odwrotna transkrypcja, w przypadku zaobserwowania amplifikacji PCR najbardziej prawdopodobne jest zanieczyszczenie DNA.
Czas postu: 02-08-2022








