1. Wykryj absorbancję roztworu RNA
Absorbancja przy 280, 320, 230 i 260 nm reprezentuje odpowiednio wartości kwasu nukleinowego, tła (mętność roztworu), stężenia soli i materii organicznej, takiej jak białko.Ogólnie patrz tylko na OD260/OD280 (stosunek, R).Gdy 1,8 ~ 2,0, uważamy, że zanieczyszczenie RNA białkiem lub inną materią organiczną może być tolerowane, ale należy zauważyć, że gdy Tris jest używany jako bufor do wykrywania absorbancji, wartość R może być większa niż 2 (ogólnie powinna wynosić <2,2).Gdy R<1,8 zanieczyszczenie białkiem lub inną materią organiczną w roztworze jest bardziej oczywiste, a los RNA można określić zgodnie z potrzebami.Gdy R>2,2 oznacza to, że RNA uległ hydrolizie do pojedynczego kwasu nukleinowego.
2.Elektroforetyczny wzór RNA
Generalnie żel denaturujący jest używany do elektroforezy RNA, ale jeśli służy tylko do wykrywania jakości RNA, żel denaturujący nie jest konieczny i można użyć zwykłego żelu agarozowego.Celem elektroforezy jest wykrycie integralności prążków 28S i 18S oraz ich stosunku, czyli integralności rozmazu mRNA.Generalnie, jeśli prążki 28S i 18S są jasne, wyraźne i ostre (w odniesieniu do krawędzi prążków są wyraźne), a jasność prążka 28S jest ponad dwukrotnie większa niż prążka 18S, jakość RNA uważamy za dobrą.
Powyższe dwie metody są powszechnie stosowane, ale żadna z tych dwóch metod nie może nam jednoznacznie powiedzieć, czy w roztworze RNA jest resztkowa RNaza.Jeśli w roztworze jest bardzo mała ilość RNazy, trudno nam ją wykryć powyższą metodą, ale większość kolejnych reakcji enzymatycznych przeprowadza się w temperaturze powyżej 37 stopni i przez długi czas.W ten sposób, jeśli w roztworze RNA będzie bardzo mała ilość RNazy, to będzie bardzo odpowiednie środowisko i czas do odegrania swojej roli w kolejnych eksperymentach, no i oczywiście eksperyment będzie wtedy zimny.Poniżej przedstawiamy metodę, która może potwierdzić obecność resztkowej RNazy w roztworze RNA.
3. Test zachowania ciepła
W zależności od stężenia próbki pobrać dwa 1000 ng RNA z roztworu RNA i dodać do 0,5 ml probówki wirówkowej, uzupełnić buforem Tris o pH 7,0 do całkowitej objętości 10 ul, a następnie szczelnie zamknąć probówkę.Umieść jedną z nich w łaźni wodnej o stałej temperaturze 70°C i trzymaj w cieple przez 1 godzinę.Drugą część przechowywano w lodówce -20°C przez 1 godzinę.Gdy czas się skończy, wyjmij dwie próbki do elektroforezy.Po zakończeniu elektroforezy porównaj pasma elektroforetyczne obu.Jeśli prążki obu próbek są spójne lub nie różnią się znacząco (oczywiście ich prążki również spełniają warunki w metodzie 2), oznacza to, że w roztworze RNA nie ma resztkowych zanieczyszczeń RNazą, a jakość RNA jest bardzo dobra.Przeciwnie, jeśli próbka inkubowana w temperaturze 70°C wykazuje wyraźną degradację, oznacza to, że w roztworze RNA występuje zanieczyszczenie RNazą.
2 Eksperymentalne metody i techniki ekstrakcji RNA
Problemy, które często napotykamy podczas ekstrakcji RNA to: (1) wydajność RNA jest niska;(2) RNA ma poważne zanieczyszczenie solą;(3) RNA ma poważne zanieczyszczenie rozpuszczalnikami organicznymi;(4) degradacja próbki i inne problemy
1. Powszechnie stosowane odczynniki do ekstrakcji całkowitego RNA
Metoda izotiocyjanianu guanidyny i metoda Trizolu to najczęściej stosowane metody ekstrakcji całkowitego RNA z tkanek i komórek zwierzęcych.Jest szczególnie odpowiedni do małych próbek i tkanek, które są szczególnie trudne do ekstrakcji, takich jak ekstrakcja całkowitego RNA ze skóry królika i zwierzęcej tkanki łącznej;ponadto Trizol, jako uniwersalny odczynnik do lizy, może być również stosowany do ekstrakcji tkanek roślinnych, bakterii, grzybów i innych tkanek.W przypadku tkanek roślinnych zawierających polisacharydy i polifenole, takich jak kamelia olejodajna, liście herbaty, nasiona rzepaku itp., metodę CTAB można również zastosować do ekstrakcji całkowitego RNA.
Jako metoda konwencjonalna metoda dwukolumnowa jest również bardzo popularna ze względu na pracę w normalnej temperaturze, brak konieczności dodawania RNazy oraz bezpieczeństwo – brak chloroformu, fenoli i innych odczynników organicznych do ekstrakcji.(rekomendowane produkty )

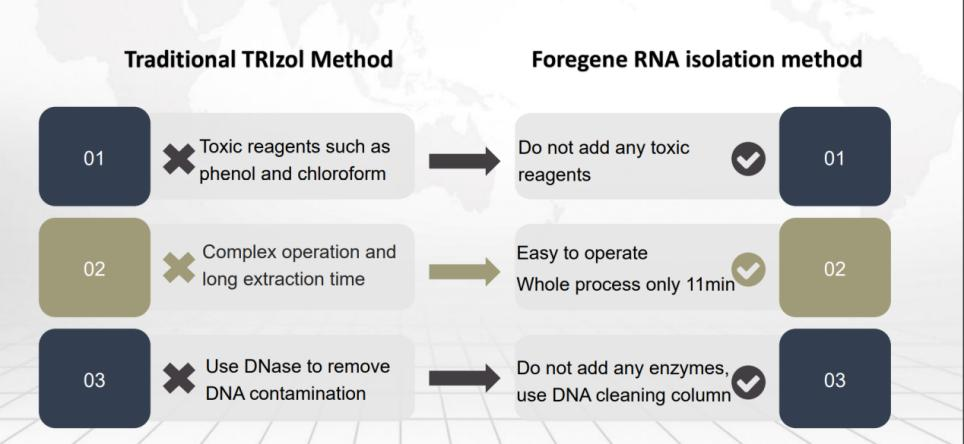
2. Ekstrakcja całkowitego RNA z tkanek zwierzęcych
(1) Spróbuj wybrać świeżą tkankę, jeśli nie jest świeża (najlepiej w ciągu trzech miesięcy - lodówka 80 ℃ lub zamrożona w ciekłym azocie. Podczas cięcia tkanki nie tnij bezpośrednio w temperaturze pokojowej, pamiętaj, aby umieścić ją w pojemniku na lód, staraj się unikać wielokrotnego zamrażania i rozmrażania.
(2) Użyj czystych nożyczek i pęsety, aby wyciąć mały kawałek tkanki, spróbuj przeciąć środkową część tkanki podczas cięcia próbki lub najpierw odetnij duży kawałek tkanki od środka, a następnie odetnij próbkę w miejscu świeżego nacięcia.Usuniętą tkankę należy całkowicie rozdrobnić, umieścić rozdrobnioną tkankę w probówce EP bez RNazy, dodać lizat, rozdrobnioną tkankę należy w pełni wystawić na działanie lizatu i przygotować do homogenizacji.
(3) W przypadku normalnych tkanek wybrać tkanki wielkości fasoli mung (30-60 mg) do homogenizacji.Jeśli tkanki zawierają dużą ilość białka, tłuszczu lub gęstej tkanki włóknistej, takiej jak wątroba, odpowiednio zwiększ lub zmniejsz ilość ciętych tkanek (opcjonalnie) Wybierz 10~20 mg).
(4) W przypadku ekstrakcji mięśni ryb, mięsa krewetek, meduz i innych tkanek o dużej zawartości wody należy odpowiednio zwiększyć objętość próbki (zalecane 100-200 mg).
(5) Jeżeli pozwalają na to warunki, tkankę zwierzęcą można pobrać bezpośrednio po homogenizacji za pomocą wysokoprzepustowego homogenizatora tkanek, jeżeli nie ma takiego sprzętu.
(6) RNA otrzymane po końcowej ekstrakcji należy natychmiast umieścić w pojemniku na lód, aby ograniczyć degradację RNA.
3. Ekstrakcja RNA z komórek zwierzęcych
(1) Zawiesina komórek: odwirować bezpośrednio i wyrzucić pożywkę, przemyć sterylnym PBS 1-2 razy, następnie zawiesić w odpowiedniej ilości PBS, a następnie dodać lizat do lizy.Nie dodawać lizatu bezpośrednio do wytrąconych komórek po całkowitym usunięciu płynu.Spowoduje to, że pakiet histonów uwolniony po lizie komórek na zewnętrznej warstwie przylgnie do zewnętrznej strony wytrąconych komórek, ograniczając w ten sposób kontakt komórek wewnątrz osadu z lizatem., powodując niepełną lizę komórek i zmniejszoną wydajność RNA.
(2) Komórki częściowo przylegające lub słabo przylegające: Po wyrzuceniu pożywki przemyć PBS 1-2 razy, następnie bezpośrednio zaabsorbować odpowiednią ilość PBS i przedmuchać szalkę hodowlaną pipetą lub pistoletem, aby zdmuchnąć komórki i przenieść je do komórek wolnych od RNA.Dodać lizat do probówki EP zawierającej enzym do ekstrakcji.
(3) Komórki przylegające: należy najpierw strawić trypsyną, następnie zebrać do probówek EP wolnych od RNaz, odwirować w celu usunięcia supernatantu, przemyć 1-2 razy PBS w celu usunięcia nadmiaru trypsyny i ponownie zawiesić w odpowiedniej ilości PBS. Następnie przejść do etapu ekstrakcji.
4. Ekstrakcja roślinnego RNA
Tkanki roślinne są bogate w związki fenolowe lub bogate w polisacharydy lub zawierają niezidentyfikowane metabolity wtórne lub mają wysoką aktywność RNazy.Substancje te po lizie komórkowej ściśle łączą się z RNA, tworząc nierozpuszczalne kompleksy lub osady koloidalne, które są trudne do usunięcia.Dlatego, gdy pobieramy tkankę roślinną, musimy wybrać zestaw do roślin.Znajdujący się w zestawie lizat skutecznie rozwiązuje problemy łatwego utleniania polifenoli oraz rozdzielania związków polisacharydowych i kwasów nukleinowych.
(Do ekstrakcji roślinnego RNA polisacharydu polifenolu zalecane produkty:
(1) Skórkę, miąższ, nasiona, liście itp. rośliny należy całkowicie zmielić w moździerzu.Podczas procesu mielenia należy na czas uzupełniać ciekły azot, aby uniknąć stopienia próbki.Zmieloną próbkę należy szybko dodać do lizatu i wstrząsnąć, aby uniknąć degradacji RNA.
(2) W przypadku próbek bogatych w błonnik, takich jak liście ryżu i pszenicy, ilość ekstrakcji powinna być odpowiednio zmniejszona, w przeciwnym razie rozdrabnianie i liza tkanki nie zostaną zakończone, co spowoduje niską wydajność wyekstrahowanego RNA.
(3) W przypadku tkanek roślinnych o dużej zawartości wody, takich jak owoc granatu, owoc arbuza, owoc brzoskwini itp., należy odpowiednio zwiększyć wielkość próbki (100-200 mg jest opcjonalne).
(4) Tkanki roślinne, takie jak liście roślin, kłącza, twarde owoce i inne materiały, są generalnie zalecane do stosowania ciekłego azotu w celu dokładnego zaprawy składników w moździerzu, a następnie przejścia do etapu ekstrakcji.Konwencjonalne homogenizatory tkanek mogą nie być skuteczne w homogenizacji tkanek roślinnych i generalnie nie są zalecane.
5. Środki ostrożności dotyczące ekstrakcji RNA
(1) Próbki tkanek powinny być jak najświeższe, aby uniknąć wielokrotnego zamrażania i rozmrażania.
(2) Tkanka powinna być całkowicie zmielona podczas ekstrakcji, a ilość tkanki nie powinna być za mała, nie mówiąc już o zbyt dużej.
(3) Po dodaniu lizatu należy zapewnić wystarczający czas inkubacji, aby w pełni zlizować próbkę.
(4) Podczas stosowania metody Trizol do ekstrakcji zasadą absorpcji supernatantu po rozwarstwieniu jest „woleć wdychać mniej niż wdychać więcej” i nie wolno ekstrahować do warstwy środkowej, w przeciwnym razie spowoduje to poważne skażenie genomowym DNA.
(5) Podczas mycia płyn do mycia powinien całkowicie przeniknąć wokół ścianki rury, aby zapewnić dokładne mycie.
(6) W przypadku metody ekstrakcji kolumny, oprócz odłączenia kolumny po umyciu, kolumnę adsorpcyjną należy również umieścić w ultra-czystej ławce i przedmuchać przez 5-10 minut, aby całkowicie odparować rozpuszczalnik organiczny do sucha.
(7) Przy ostatniej elucji metody kolumnowej, po dodaniu wody DEPC, należy ją inkubować przez 3-5 minut lub wcześniej podgrzać wodę DEPC do 60°C, aby zwiększyć wydajność elucji.W tradycyjnej metodzie cięcia Trizolem i wytrącania izopropanolem końcowe RNA rozpuszcza się w wodzie DEPC, dlatego należy zapewnić odpowiedni czas na rozpuszczenie, a dno probówki wirówkowej należy w sposób ciągły przedmuchać końcówką pipety.
3 Three Przyczyny i rozwiązania dla niskiego stężenia RNA/złej jakości
1. Wydajność jest zbyt niska
Wyekstrahowana próbka jest za mała, całkowita ilość jest niewystarczająca lub wyekstrahowana próbka jest za duża i liza nie jest zakończona;do ekstrakcji należy użyć tkanki lub komórek odpowiedniej jakości, wstępne przygotowanie próbki musi być dobrze wykonane, a liza powinna być wystarczająca.
2. Pozostałości genomu
W przypadku ekstrakcji metodą Trizol, gdy supernatant jest zasysany do środkowej warstwy po nałożeniu warstw, spowoduje to poważne zanieczyszczenie genomu;podczas układania warstw należy zachować szczególną ostrożność, aby uniknąć zasysania do środkowej warstwy.Jeżeli do ekstrakcji stosowana jest metoda kolumnowa, do ekstrakcji można wybrać zestaw zawierający DNazę I.Kwas nukleinowy zaadsorbowany na membranie jest bezpośrednio trawiony DNazą I, co może znacznie zredukować pozostałości DNA.
3. Degradacja RNA
Może to być degradacja samej wyekstrahowanej próbki lub degradacja spowodowana procesem ekstrakcji;w miarę możliwości do ekstrakcji RNA należy używać świeżych próbek, a pobrane próbki przechowywać w ciekłym azocie lub lodówce -80°C na czas i unikać wielokrotnego zamrażania i rozmrażania.W procesie ekstrakcji RNA należy używać końcówek wolnych od RNaz/DNaz, probówek wirówkowych i innych materiałów.Proces ekstrakcji powinien przebiegać tak szybko, jak to możliwe.Wyekstrahowany RNA należy umieścić w pojemniku z lodem i przechowywać w temperaturze -80°C.Jeżeli wyekstrahowany RNA musi być wykryty metodą elektroforezy żelowej, elektroforezę należy wykonać natychmiast po ekstrakcji, a bufor do elektroforezy wymienić na nowo przygotowany.
4. Pozostałości soli i rozpuszczalników organicznych
Odczynniki do ekstrakcji zawierają sole fenolu i guanidyny, a roztwór płuczący zawiera etanol.Podczas procesu ekstrakcji lizat nie został całkowicie wchłonięty i odrzucony, a roztwór płuczący nie został całkowicie wysuszony.Resztkowe sole i rozpuszczalniki organiczne są szkodliwe dla późniejszej odwrotnej transkrypcji i PCR.Różne stopnie inhibicji, dlatego lizat tkankowy powinien być całkowicie usunięty podczas procesu ekstrakcji, a przemycie powinno być wystarczające, aby można było przemyć otaczające ścianki probówki.Ponadto rura jest opróżniana i przedmuchiwana, co jest niezbędnym krokiem, co dodatkowo zmniejszy pozostałości materii organicznej.
Więcej informacji na temat ekstrakcji RNA można znaleźć na naszej stronie internetowej:
www.foreivd.com, aby uzyskać więcej informacji.

Czas postu: grudzień-01-2022